Die Erforschung der komplexen molekularen Verteilung im Gewebe stellt einen zentralen Bereich der modernen Biowissenschaften dar. Besonders das Verständnis, wie Biomoleküle wie Lipide, Metaboliten, Proteine und Glykane räumlich innerhalb von Geweben organisiert sind, bietet wichtige Einblicke in physiologische Prozesse und Krankheitsmechanismen. Eine innovative Technik, die zunehmend an Bedeutung gewinnt, ist die Massenspektrometrie-Bildgebung (MSI), welche molekulare Signaturen im Gewebe ohne Aufzeichnung von Markern sichtbar macht. Allerdings war die bisherige Anwendung dieser Technologie durch limitierte räumliche Auflösung und entweder sehr langsame Messzeiten oder hohe Kosten eingeschränkt. Hier setzt TEMI – Tissue-Expansion Mass-Spectrometry Imaging – an und stellt eine zukunftsweisende Lösung dar, welche nicht nur hochauflösende molekulare Bilder ermöglicht, sondern dabei kosteneffizient und anpassbar bleibt.
TEMI kombiniert Gewebeexpansion mit MALDI-Massenspektrometrie und erlaubt so das Abbilden von Biomolekülen im mikrometergenauen Detail. Das Prinzip der Gewebeexpansion beruht darauf, dass Gewebe durch chemisch verankerte Polymernetzwerke in ein hydrogelartiges Material eingebettet und anschließend mit Wasser aufgequollen werden. Dies vergrößert die Gewebestruktur linear um ein Vielfaches, ohne dabei ihre molekulare Architektur zu zerstören. Klassische Expansionsmethoden arbeiten meist mit hohen Temperaturen, Detergenzien und enzymatischer Verdauung, was zwar die Expansion unterstützt, jedoch zu Verlust oder Veränderung wichtiger Biomoleküle führen kann. Im Gegensatz dazu verzichtet TEMI auf proteolytischen oder thermischen Denaturationsschritte und bewahrt somit die native Verteilung von Proteinen, Lipiden und kleinen Molekülen besser – eine entscheidende Voraussetzung für präzise Massenspektrometrie-Bildgebung.
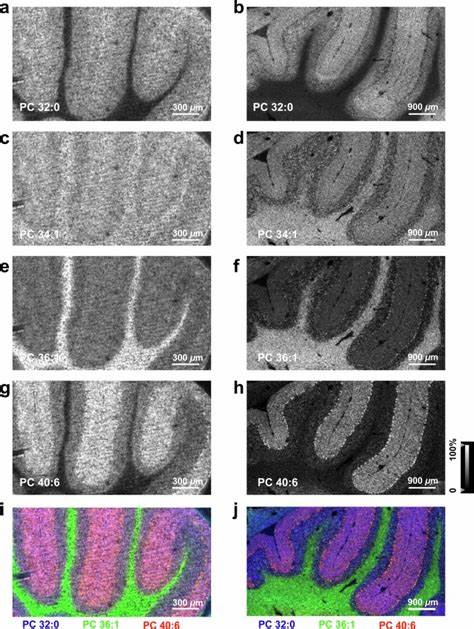
Die physikalische Vergrößerung des Gewebes sorgt für eine effektive Verbesserung der räumlichen Auflösung bei der MSI. Konkret bedeutet eine fingerbreite Vergrößerung von etwa 2,5- bis 3,5-fach, dass Messungen mit einem Laser-Raster von 50 µm oder sogar 10 µm eine vergleichbare Auflösung wie 20 µm bzw. 3 µm im ursprünglichen, nicht expandierten Gewebe erzielen. Dies ermöglicht erstmals die Analyse molekularer Verteilungen auf Single-Cell-Ebene ohne die üblichen technischen und finanziellen Hürden, die mit teuren Hochauflösungssystemen verbunden sind. Ein Beispiel für die Leistungsfähigkeit von TEMI liefert die Untersuchung des Maus-Cerebellums.
Mittels TEMI können verschiedene Zellschichten des Kleinhirns präzise kartiert werden, wobei unterschiedliche Lipidspezies klar in den funktionell differenzierten Schichten wie der Molekular-, Granulär- und Weißsubstanzschicht lokalisiert werden. So lassen sich Phosphatidylcholine wie PC (32:0), PC (38:1) oder PC (38:6) als molekulare Marker für die unterschiedlichen Zonen nachweisen. Durch Steigerung der Expansionsfaktor und Reduzierung der Laser-Rastergröße können sogar einzelne Purkinje-Zellen abgebildet und analysiert werden. Diese Auflösung war mit traditionellen MSI-Technologien ohne Expansionsverfahren nicht praktikabel. Neben Lipiden gelingt es TEMI auch, wichtige Metaboliten, Proteine und N-Glykane im Gewebe zu erfassen.
Die Methode verwendet verschiedene MALDI-Matrizen und Detektionsmodi, um zelluläre Metaboliten wie Cholin, Hexose-Phosphate, Oleoylethanolamid oder 2-Arachidonylglycerol räumlich abzubilden. Proteine werden in situ durch enzymatischen Trypsin-Digest freigesetzt und mittels MALDI-MS sequenziert. Dabei konnten wichtige Proteine wie das Myelin Basic Protein sowie Histone in unterschiedlichen Gewebeschichten lokalisiert werden. Die Erfassung von N-Glykankarten, die für Zellkommunikation und -adhäsion eine maßgebliche Rolle spielen, ergänzt die molekulare Analyse und beleuchtet glykan-spezifische Muster innerhalb der Gewebearchitektur. Neben gesunden Organen wurde TEMI auch auf Tumorgewebe angewendet.
Die Methode konnte die metabolische Heterogenität in Melanomgewebe deutlich besser auflösen als herkömmliche MSI. Mit einem 3,5-fach expandierten Gewebe ließen sich wesentlich mehr unterschiedliche Cluster molekularer Signaturen segmentieren, was die komplexe Biologie von Tumorzellen im Mikroumfeld minutiös enthüllt. Diese Fähigkeit eröffnet neue Wege für die Krebsforschung, sowohl hinsichtlich der Diagnose als auch zur Identifikation von Therapieansätzen, die molecular differenziert und räumlich fokussiert sind. Das Protokoll für TEMI ist neben dem Kleinhirn und Tumorgewebe bereits erfolgreich auf diverse Organe wie Niere und Bauchspeicheldrüse anwendbar, mit vergleichbarer Bildqualität und minimalen Gewebeverzerrungen im Bereich unter 12 Prozent. Diese Vielseitigkeit zeigt das Potenzial von TEMI als breit einsetzbares Werkzeug für räumliche Multi-Omics-Analysen in der biomedizinischen Forschung.
Technische Herausforderungen wurden im Rahmen der Entwicklung von TEMI adressiert: So wurde die Kryoschnitttechnik optimiert, um stabile, dünne Gewebeschnitte mit Homogenität zu gewährleisten. Die doppelte bis dreifache Gelierung und Expansion erhöht dabei die mechanische Stabilität des expandierten Gewebes und minimiert Gewebeverzerrungen und Rissbildungen. Außerdem wurden unterschiedliche Matrizen und Behandlungsschritte so angepasst, dass eine optimale Ionisation der unterschiedlichen Biomoleküle erreicht wird. Eine kritische Neuerung von TEMI liegt im Verzicht auf proteinspaltende oder harsche Denaturierungsprozeduren, was die Analytenintegrität bewahrt. TEMI ist kompatibel mit verschiedenen Massenspektrometrie-Bildgebungsplattformen, vorwiegend mit MALDI-MSI, kann aber auch zukünftig mit Desorptions- oder Lasermikrodispersionsverfahren weiterentwickelt werden.
Die Kombination mit Immunhistochemie anhand photocleavbarer Massentags erweitert die Möglichkeiten zur multiplexen Protein-Dekodierung in Gewebeschnittbildern. Trotz aller Fortschritte weist TEMI auch Limitationen auf. Die Dichte von Biomolekülen wird mit zunehmender Expansion geringer, was die Empfindlichkeit der Massenspektrometrie herausfordert. Besonders bei kleinen, frei löslichen Molekülen könnte es während der Wassereinlagerung zu Diffusionsverlusten kommen, die die räumliche Genauigkeit reduzieren. Weitere chemische Verankerungsmethoden für diese Substanzen könnten dieses Problem mindern.
Auch die Ausdehnung in der Z-Achse bleibt durch Schnitttiefe limitiert, was dreidimensionale Bildgebung momentan erschwert. Die Perspektiven von TEMI sind dennoch vielversprechend. Das Verfahren kann noch weiter optimiert werden, um neben Lipiden, Metaboliten und Proteinen auch andere Molekülklassen beispielsweise Nukleinsäuren oder Lipid-gebundene Signalmoleküle räumlich immer feiner darzustellen. Die mögliche zehnfache Expansion, die aktuell experimentell angestrebt wird, könnte Auflösungen im Submikrometerbereich realisierbar machen. Darüber hinaus eröffnet die Kombination von TEMI mit weiteren analytischen Verfahren, wie optischer Mikroskopie, Einzelzellsequenzierung oder funktioneller Bildgebung, völlig neue multidimensionale Blickwinkel auf die molekulare Gewebegranularität.
Abschließend lässt sich zusammenfassen, dass TEMI durch die innovative Kombination von schonender Gewebeexpansion und hochauflösender Massenspektrometrie ein leistungsstarkes Werkzeug für die molekulare Gewebekartierung ist. Die Methode macht die Einzelzellauflösung von Biomolekülen in intakten Organen zugänglich und ermöglicht ein besseres Verständnis von biologischen Prozessen und pathologischen Veränderungen wie Tumorheterogenität. Seit der Veröffentlichung der Methode im Jahr 2025 hat sich TEMI als praktikables, flexibles und vergleichsweise kostengünstiges Verfahren etabliert, das vielfältige Anwendungspotenziale sowohl in der Grundlagenforschung als auch in der klinischen Diagnostik bietet. Die Expansion der räumlichen Omics-Technologien durch TEMI ebnet den Weg für präzisere, personalisierte und molekular fundierte medizinische Ansätze der Zukunft.