Die Frage danach, wie Moleküle zu Zeichen werden und welche Mechanismen biologische Information ermöglichen, zählt zu den faszinierendsten Herausforderungen moderner Wissenschaft. Seit Jahrzehnten dominieren Vorstellungen, wonach DNA und RNA die eigentlichen Träger biologischer Information sind. Diese Sichtweise, oft durch das zentrale Dogma der Molekularbiologie verkörpert, setzt voraus, dass Moleküle wie DNA und RNA intrinsisch Bedeutungen oder Information besitzen. Doch diese Perspektive greift zu kurz, wenn es darum geht, die tatsächliche Bedeutung dessen zu erfassen, was biologische Information in lebenden Systemen ausmacht. Ein grundlegender Wendepunkt in unserem Verständnis von biologischer Information liegt in der Erkenntnis, dass Information nicht einfach in Molekülsequenzen steckt, sondern dass sie erst durch komplexe interpretative Prozesse real wird.
Moleküle werden zu Zeichen nicht durch ihre bloße physikalisch-chemische Beschaffenheit, sondern durch Prozesse, die ihre Eigenschaften referenziell und funktional nutzen. In diesem Sinne ist biologische Information immer semantisch eingebettet und benötigt einen »Interpreter«, der die molekulare ›Botschaft‹ liest, deutet und für den Organismus nutzbar macht. Historisch gesehen lieferte Erwin Schrödinger mit seinem Werk »Was ist Leben?« (1944) eine wegweisende physikalische Perspektive auf Organismen als Systeme, die fern vom thermodynamischen Gleichgewicht bleiben und Informationen speichern, welche ihre Organisation steuern. Schrödingers Konzept des »aperiodischen Kristalls« als Informationsträger inspirierte die Entdeckung der DNA-Doppelhelix und trug wesentlich zu der damals neuen biologischen Informationsauffassung bei. Im Anschluss definierte Francis Crick das zentrale Dogma, das DNA als Informationsquelle betrachtet, deren Sequenzen zu RNA und Proteinen abgelesen werden.
Dieser materialistische Ansatz reduzierte biologische Information auf rein physikalische Muster, bei denen Interpretation als einfaches Kopieren verstanden wurde. Dieses Denken wurde populär durch Richard Dawkins’ »egoistisches Gen«, das Information als Replikation kodierter Molekülsequenzen definierte. Doch diese Sichtweise übersah die Frage nach der Bedeutung, der sogenannten »Aboutness« biologischer Information. Physikalische Muster allein sind nicht ›über etwas‹, sondern erhalten ihre Bedeutung erst innerhalb eines geeigneten semantischen Kontextes durch Mechanismen der Interpretation. Die Grenzen dieses reinen Replikationsmodells zum Verständnis biologischer Bedeutung werden deutlich, wenn man sich den Ursprung des Lebens anschaut.
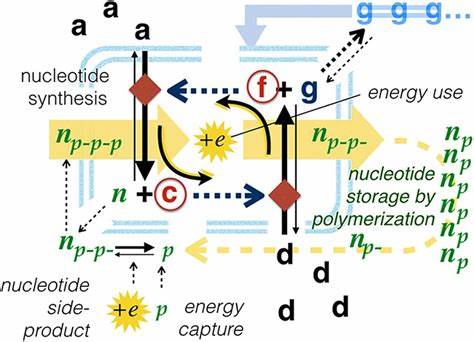
Ursprüngliche Lebensformen mussten einfache molekulare Systeme sein, die dennoch interpretative Kompetenzen besaßen – also in der Lage waren, funktionale Beziehungen zur Umwelt herzustellen, Informationen zu re-präsentieren und diese zur Aufrechterhaltung ihrer Organisation zu nutzen. Solche Basissysteme bewegten sich fern vom thermodynamischen Gleichgewicht und konnten Selbstreparatur und Selbstreproduktion vollziehen. Ein interessantes Modell hierfür sind sogenannte »Autogene«, hypothetische molekulare Gebilde, die auf der Kopplung zweier zentraler Prozesse basieren: der reziproken Katalyse und der Selbstassemblierung. Reziproke Katalyse beschreibt einen Kreislauf, in dem eine katalytische Reaktion Produkte generiert, die weitere Reaktionen katalysieren, wobei eine zirkuläre Beziehung entsteht, welche die Produktion von Molekülen aufrechterhält. Selbstassemblierung wiederum ermöglicht es bestimmten Molekülen, sich spontan zu geordneten Strukturen zusammenzufügen – wie es bei Viruscapsiden oder Zellmembranen beobachtet wird.
Die Wechselwirkung zwischen diesen beiden Prozessen schafft Bedingungen, bei denen sich komplexe molekulare Systeme stabilisieren und sich dynamisch weiterentwickeln können. Ein Autogen kann sich durch diese Mechanismen selbst reparieren und sogar selbst reproduzieren, wobei es auf lokale Umweltbedingungen reagiert und seine Aktivitäten an die Verfügbarkeit von Substraten koppelt. So zeigt sich hier bereits ein rudimentäres interpretatives Verhalten, in dem molekulare Veränderungen innerhalb eines dynamischen Systems als bedeutungsvoll angesehen und in selbstorganisierenden Abläufen berücksichtigt werden. Diese selbstaufrechterhaltenden molekularen Zyklen bieten eine Grundlage für biologische Information, die nicht mehr nur als eine Eigenschaft des Moleküls verstanden wird, sondern als emergente Eigenschaft eines Systems, das Interpretationskompetenz besitzt. Die Kernidee ist, dass Information auf Einschränkungen (Constraints) beruht, die den Ablauf physikalischer Prozesse lenken und lenkbar machen – also auf strukturellen Bedingungen, die die Ausführung von Arbeit ermöglichen, ohne dabei selbst verbraucht zu werden.
Hier kommt auch der Begriff der »semiotischen Affordanzen« ins Spiel, der beschreibt, wie die physikalischen Eigenschaften von Molekülen Potenziale für bestimmte Interpretationen bieten, die jedoch erst im Zusammenspiel mit einem interpreterenden System Wirklichkeit werden. Das bedeutet, dass eine Eigenschaft eines Moleküls nicht automatisch eine bestimmte Bedeutung trägt, sondern dass Bedeutung immer durch den Kontext der Interpretation erzeugt wird. Ein bedeutender Schritt auf dem Weg zur Entstehung komplizierter biologischer Information ist die Verschiebung von dynamischen Wechselwirkungen auf statische molekulare Strukturen – die sogenannte »offloading« oder Verlagerung von Constraints. Polynukleotide wie DNA und RNA dienen hier als stabile Substrate, auf denen dynamische molekulare Interaktionen gespeichert und instruierend wiedergegeben werden können, ohne selbst durch die Dynamik verändert zu werden. Diese Verlagerung ermöglicht eine Form der Repräsentation, in der sich das System selbst referenziert und seine eigenen Voraussetzungen reproduziert – eine Art semantische Rekursion.
Freeman Dyson hatte bereits in den 1980er Jahren den Hinweis gegeben, dass Nucleotide nicht ausschließlich als Informationsmoleküle entstanden sind, sondern auch energetische Funktionen innehatten. Ihre Rolle als Energieträger (z. B. ATP) könnte evolutionär älter sein als ihre Rolle als Informationsspeicher. Die schrittweise Rekrutierung von Nukleotiden für Informationsfunktionen ist ein plausibles Szenario, das experimentell und theoretisch weiter verfolgt wird.
Die Verschiebung der Interaktionsmuster von katalytischen Proteinen auf Nukleinsäurepolymere bietet den Vorteil, dass aufgrund der relativ stabilen Sequenzen von DNA oder RNA komplexe molekulare Netzwerke effizient gesteuert und reguliert werden können. Dies umgeht die »kombinatorische Katastrophe«, die bei der Vielzahl möglicher zufälliger molekularer Interaktionen problematisch wird. Durch die regulatorische Rolle von Nukleinsäuren kann die molekulare Komplexität gesteigert und die Evolvierbarkeit von Lebewesen insgesamt deutlich erhöht werden. Aus biosemiotischer Sicht stellt dieser Prozess einen entscheidenden Entwicklungsschritt dar, weil hier erstmals physikalisch zugängliche Zeichenmedien geschaffen wurden, die eine systemeigene Bedeutung und Funktion besitzen. Die klassische Peirce’sche Dreiteilung von Zeichen zu Interpretant und Objekt lässt sich hier in molekularen Prozessen abbilden: Moleküle fungieren als Signale (Representamen), die durch die Systemdynamiken interpretiert werden und auf Umweltbedingungen oder interne Zustände (Objekte) verweisen.
Erweiterungen der autogenen Modelle zeigen, dass komplexere semiotische Funktionen möglich sind. Beispielsweise können molekulare Systeme sensitiv auf Umweltbedingungen reagieren, indem Capsidstrukturen spezifische Bindungsstellen für Substrate anbieten, deren Konzentration den Zerfall und die Freisetzung katalytischer Komponenten reguliert. So wird eine Art indexikalische Interpretation möglich, bei der molekulare Veränderungen direkt mit Umweltvariablen korreliert werden. Diese molekularen Basiskompetenzen legen die Struktur biologischer semiotischer Hierarchien frei. Über sogenannte »semiotische Gerüste« (semiotic scaffolding) entsteht eine Rekursion höherer Ordnungen, die sich von molekularen Prozessen über genetische Regulation bis hin zu zellulären, geweblichen und organismischen Ebenen erstrecken.
So können komplexe Organismen entstehen, deren Entwicklungs- und Verhaltensmuster durch verschachtelte Zeichenprozesse organisiert sind. Zusammenfassend lässt sich feststellen, dass die traditionelle Auffassung, wonach Moleküle wie DNA oder RNA allein die Quelle biologischer Information seien, einer Neubewertung bedarf. Vielmehr sollte Information als eine Eigenschaft dynamischer Systeme verstanden werden, die interpretative Fähigkeiten besitzen. Moleküle werden erst durch solche interpretativen Prozesse zu Zeichen, deren Bedeutung über die rein materiellen Eigenschaften hinausgeht. Die Entstehung des Lebens, seiner Informationssysteme und damit des biologischen Sinns ist somit eng verknüpft mit den evolutionären Entwicklungen selbstorganisierender, selbstrepräsentierender molekularer Systeme, die kontinuierlich Signale erzeugen, deuten und verwenden.
Dieser Paradigmenwechsel hat weitreichende Konsequenzen nicht nur für die Biologie, sondern auch für die Philosophie der Information, Chemie, und kognitive Wissenschaften. Er eröffnet neue Wege, um den Ursprung des Lebens, die Entstehung von Bedeutung, und die Natur von Information aus einem holistischen, systemischen und semiotischen Blickwinkel zu untersuchen. Die Herausforderung liegt nun daran, diese theoretischen Einsichten empirisch und experimentell auszugestalten, um so unser Verständnis von Leben und seinen Ursprüngen weiter zu vertiefen.