Die Entdeckung und Analyse von Viren in biologischen Proben hat in den letzten Jahren durch neue technologische Innovationen einen bedeutenden Fortschritt erfahren. Eine der faszinierendsten Entwicklungen ist die Einführung benutzerfreundlicher Software, die Viren in RNA-Sequenzdaten erkennen kann. Diese Fortschritte sind vor allem für die molekulare und computergestützte Biologie von großer Bedeutung, da sie es Forschern ermöglichen, aus komplexen RNA-Datensätzen wertvolle Informationen über Viren zu extrahieren, die bislang oft unbeachtet blieben. Virenerkennung in RNA-Sequenzen: Eine Herausforderung RNA-Viren sind verantwortlich für viele der häufigsten und auch schwerwiegenden Infektionskrankheiten weltweit. Trotzdem war die Identifizierung viraler RNA in gemischten Proben, die neben viraler RNA auch Wirts-RNA enthalten, lange Zeit eine anspruchsvolle Aufgabe.
In einem typischen RNA-Sequenzierungsdatensatz dominieren die genetischen Informationen des Wirtes, wodurch virale Sequenzen oft übersehen oder ausgesondert werden. Daraus resultiert, dass wichtige Hinweise auf virale Infektionen oder die Rolle von Viren in biologischen Prozessen verloren gehen. Moderne Sequenziertechnologien wie die Einzelzellanalyse (Single-Cell RNA-Seq) erzeugen enorme Datenmengen, die zwar detaillierte Einblicke in die Genexpression individueller Zellen bieten, aber auch eine Herausforderung für die Datenanalyse darstellen. Virale RNA in solchen Daten zu identifizieren, benötigt deshalb nicht nur hochentwickelte bioinformatische Methoden, sondern auch nutzerfreundliche Software, die auch Wissenschaftlern ohne tiefgehende Computerkenntnisse den Weg zu diesen Erkenntnissen ebnet. Die Rolle der Software kallisto Ein wichtiger Meilenstein in diesem Bereich ist die softwarebasierte Methode kallisto, die an der California Institute of Technology entwickelt wurde.
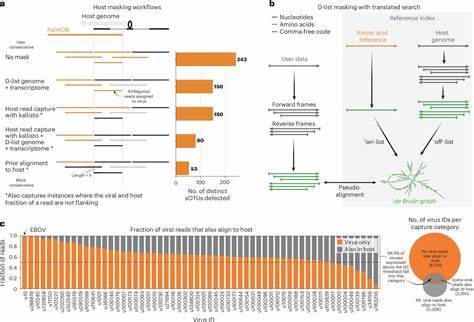
Das Programm wurde ursprünglich für die schnelle und genaue Quantifizierung von Transkriptomdaten konzipiert und wurde nun um eine Funktion erweitert, die speziell die Erkennung viraler RNA ermöglicht. Dabei liegt ein Fokus auf der Erkennung von RNA-abhängiger RNA-Polymerase (RdRp), einem essentiellen Protein, das in einer Vielzahl von RNA-Viren vorkommt. Durch die gezielte Suche nach dem genetischen Fingerabdruck der RdRp kann die Software mehr als 100.000 verschiedene Virusarten identifizieren. Dies geschieht mit vergleichsweise geringem Rechenaufwand, was die Zugänglichkeit und Anwendungsmöglichkeiten stark erhöht.
Die Integration eines umfangreichen Datenbankbestandes, der als PalmDB bekannt ist, bildet dabei das Fundament für die virale Identifikation. Anwendungsgebiete und wissenschaftliche Bedeutung Die Möglichkeit, Viren in RNA-Sequenzdaten einfach zu erkennen, hat weitreichende Auswirkungen. Zum einen wird das Monitoring von neu auftretenden Krankheitserregern erheblich erleichtert. Gerade bei Epidemien oder bei der Überwachung von potenziellen Pandemien kann diese Technologie den entscheidenden Zeitvorteil bieten, indem sie frühzeitig neue Virusstämme oder unbekannte Viren entdeckt. Darüber hinaus sind viele Viren in ihrem Einfluss auf den menschlichen Organismus weitgehend unerforscht.
Es wird mittlerweile angenommen, dass einige neurodegenerative Erkrankungen wie Alzheimer oder Parkinson durch virale Infektionen ausgelöst oder beeinflusst werden könnten. Die neue Software unterstützt Forscher dabei, diese Zusammenhänge besser zu verstehen, indem sie virale RNA direkt in biologischen Proben nachweisen und quantifizieren können. Die Kombination mit Einzelzellanalyse ermöglicht es zudem, Viren nicht nur in Proben zu erkennen, sondern auch genau zu bestimmen, in welchen Zelltypen sie sich befinden und wie sie dort die Genexpression des Wirts verändern. Auf diese Weise lassen sich tiefere Einblicke in Virus-Host-Interaktionen gewinnen, die für die Entwicklung neuer Therapien und Präventionsstrategien essenziell sind. Benutzerfreundlichkeit und Zugänglichkeit Ein zentrales Merkmal der neuen Software ist ihre Benutzerfreundlichkeit.
Viele wissenschaftliche Softwarelösungen im Bereich der Bioinformatik setzen umfassende Programmierkenntnisse oder Erfahrung mit komplexen Betriebssystemen voraus, was insbesondere für Biologen eine große Hürde darstellt. Kallisto wurde mit dem Ziel entwickelt, für jedermann zugänglich zu sein, sodass Forscher mit unterschiedlichstem technischen Hintergrund die Vorteile der Software nutzen können. Dies bedeutet, dass Anwender ihre RNA-Sequenzdaten einfach in das Programm eingeben und ohne großen technischen Aufwand analysieren können. Dadurch wird ein breiteres Spektrum an Forschern erreicht, von Laborwissenschaftlern bis hin zu Klinikern, was die Verbreitung und Nutzung der Technologie fördert. Ausblick und zukünftige Entwicklungen Die rasante Weiterentwicklung in der Bioinformatik und Sequenzierungstechnik macht es wahrscheinlich, dass Software wie kallisto in den kommenden Jahren noch leistungsfähiger wird.
Neue Algorithmen könnten etwa die Erkennung von Virusmutationen verbessern oder die Entdeckung noch unbekannter Virenarten erleichtern. Auch die Integration in klinische Diagnostiksysteme ist denkbar und könnte in Zukunft helfen, Infektionskrankheiten schneller und präziser zu diagnostizieren. Zudem erlaubt der Fortschritt in der single-cell Sequenzierung ein immer detaillierteres Bild der biologischen Systeme, in denen Viren agieren. Dies eröffnet neue Forschungsfelder und könnte helfen, bislang unerklärte Krankheitsmechanismen aufzudecken. Schließlich trägt die Verfügbarkeit offener, benutzerfreundlicher Software entscheidend zur Demokratisierung der Wissenschaft bei.