Die räumliche Verteilung von Biomolekülen in Geweben ist von entscheidender Bedeutung für das Verständnis vielfältiger biologischer und pathologischer Prozesse. Doch die detaillierte Kartierung dieser Moleküle stellt Forschende vor technische Herausforderungen. Ein Durchbruch in diesem Bereich ist die Entwicklung von TEMI – Tissue-Expansion Mass-Spectrometry Imaging. Diese Methode kombiniert die aus der Expansionsmikroskopie bekannte Gewebeexpansion mit der leistungsfähigen Massenspektrometrie-Bildgebung (MSI), um die molekulare Landschaft in biologischem Gewebe mit noch nie dagewesener Auflösung und Sensitivität zu erfassen.Die Grundlage von TEMI besteht darin, Gewebeproben chemisch in ein Hydrogel einzubetten und sie dann kontrolliert zu vergrößern.
Die Expansion wird dabei so durchgeführt, dass die molekularen Strukturen weitestgehend erhalten bleiben. Klassische Gewebeexpansionsverfahren verwenden häufig proteolytische Enzyme oder chemische Denaturierung, was jedoch die Integrität bestimmter Biomoleküle beeinträchtigt und zu Verlusten oder Verlagerungen führen kann. TEMI verzichtet bewusst auf diese harten Behandlungsschritte. Stattdessen werden Proteine auf natürliche Weise in das Gel eingebunden und ohne Denaturierung expandiert. Dies ermöglicht den Erhalt von Lipiden, Metaboliten, Peptiden und N-Glykanen in einem nativen Zustand und erhöht die Erfassungsgenauigkeit der MSI deutlich.
Ein weiterer Vorteil von TEMI ist die optionale mehrfache Gelierung, durch die ein interpenetrierendes Gelnetzwerk entsteht. Dadurch wird die Stabilität und Robustheit der expandierten Probe verbessert, die Expansion erfolgt isotrop, das heißt nahezu gleichmäßig in alle Richtungen, und die Auflösung steigt beträchtlich an. Erste Anwendungen zeigten eine lineare Expansion des Gewebes um das Zwei- bis Dreifache. Das führt dazu, dass eine Laserstrahlgröße von 50 Mikrometern effektiv auf etwa 20 Mikrometer verkleinert wird, was schon hervorragende Details bis zur Einzelzellauflösung ermöglicht. Noch feinere Strukturen wurden mit bis zu dreifacher Expansion und engeren Laser-Rasterabständen sichtbar.
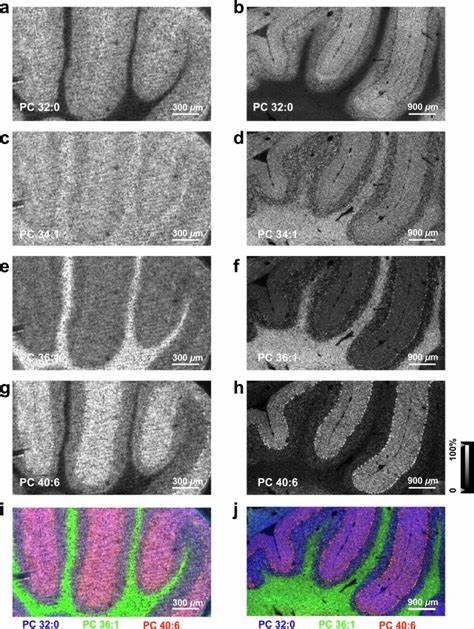
So wurden unter anderem einzelne Purkinje-Zellen im Kleinhirn mit ihren spezifischen molekularen Signaturen differenziert.Die Massenspektrometrie-Bildgebung selbst erfolgt durch Matrix-unterstützte Laserdesorptions-/Ionisation (MALDI), ein Verfahren, das biomolekulare Scans im Gewebe ohne Markierung erlaubt. Im Kombination mit TEMI bedeutet dies eine gestärkte Erfassung kleiner Moleküle, wobei gleichzeitig die Laserenergie und Probenstabilität optimiert wurden, um die biomolekulare Integrität während des Messvorgangs zu schützen. Durch die Expansion verteilt sich das Material auf eine größere Fläche, was erlaubte, bei gleichbleibender Scanzeit mehr Voxel zu erfassen. Somit profitieren Forscher von einem guten Gleichgewicht zwischen hoher räumlicher Auflösung und relativ hoher Durchsatzrate.
Zur Validierung von TEMI wurden verschiedene murine Organe untersucht, darunter das Kleinhirn, die Niere, die Bauchspeicheldrüse und Tumorgewebe. Vor allem das Kleinhirn zeigte eine differenzierte Verteilung von Phosphatidylcholin-Lipidarten in den funktionalen Schichten. Diese wurden mit TEMI deutlicher sichtbar als mit herkömmlichen MSI-Methoden. Außerdem konnten spezifische Stoffwechselprodukte wie Cholin oder Oleoylethanolamid räumlich zugeordnet werden. Diese metabolische Heterogenität dokumentiert den hohen Aussagewert der Methode.
Die Analyse wurde unter anderem auch auf Peptide und Proteine ausgeweitet. Durch in-situ Trypsin-Digestion konnten Peptide für die Massenspektrometrie aufgelöst und lokalisiert werden. Bekannte Biomarker wie das Myelin-Grundprotein oder Histone wurden mit charakteristischen Musterverteilungen abgebildet. Zusätzlich erlaubte die Methode das Mapping von N-Glykanen, einer wichtigen Klasse von Zuckerstrukturen auf Proteinen, die für viele biologische Prozesse essenziell sind.Größere Bedeutung hat TEMI insbesondere bei der Untersuchung von Tumorgeweben.
Klassischerweise leiden Analysen an fehlender molekularer Auflösung oder verpixelten Darstellungen. TEMI ermöglicht dagegen die Auflösung zahlreicher molekularer Subtypen innerhalb eines Tumors und zeigt deren heterogene Verteilung auf. In einer Studie mit einem Mausmelanom wurden 21 unterschiedliche molekulare Regionen mittels Spektren-Kluftung identifiziert, verglichen mit nur drei Clustern in einem herkömmlich analysierten nicht expandierten Tumor. Dies unterstreicht das Potenzial von TEMI, räumliche Heterogenität in der Tumormikroumgebung präzise zu erfassen und damit Erkenntnisse für Diagnose und Therapie zu liefern.TEMI ist zudem nicht nur auf ein Organ oder eine Molekülklasse begrenzt, sondern breit adaptierbar.
Die Kompatibilität mit verschiedenen MS-Modalitäten, von MALDI bis zu Laserablation oder Desorption-Elektrospray, erweitert die Anwendungsmöglichkeiten deutlich. Zudem lässt sich TEMI mit correlative Imaging-Techniken kombinieren, um molekulare und morphologische Informationen zusammenzuführen.Trotz der überzeugenden Vorteile gibt es Herausforderungen. Die Expansion führt zu einer Verdünnung der Biomoleküle, was die Sensitivität beeinträchtigen kann. Ebenfalls können sehr kleine, frei lösliche Moleküle während der Expansion diffus oder verloren gehen.
Hier ist die Entwicklung von neuen chemischen Verankerungsstrategien gefragt. Des Weiteren muss die Probenvorbereitung für möglichst reproduzierbare Mengen und minimale Deformationen weiter optimiert werden.Nicht ausgeschlossen ist die Erhöhung des Expansionfaktors über weitere Gelierungsrunden hinaus. Erste Tests deuten an, dass sogar eine etwa zehnfache lineare Expansion möglich sein könnte, was feinsten molekularen Strukturen auf Subzellulärebene zugänglich machen würde. Dies erfordert allerdings noch Balance zwischen Detailgenauigkeit und Signalstärke.
Im medizinischen Kontext eröffnet TEMI ganz neue Perspektiven. Die räumliche Multi-Omics Kartierung – also die gleichzeitige Erfassung von Lipiden, Proteinen, Metaboliten und Glykosylierungen innerhalb derselben Probe – unterstützt das Verständnis komplexer Krankheiten. Beispielsweise könnten Tumormikroumgebungen besser charakterisiert, neurologische Erkrankungen differenzierter analysiert und personalisierte Therapien zielgenauer entwickelt werden.Nicht zuletzt ist TEMI auch wirtschaftlich und technisch attraktiv. Im Vergleich zu hochspezialisierten Gerätelösungen zur MSI mit extrem hohen Kosten benötigt TEMI keine üppige Spezialhardware, sondern kann an bestehenden MALDI-MSI-Instrumenten adaptiert werden.
Die Anwendung setzt keine aufwändige Proteolytische Behandlung voraus, sondern basiert auf einem chemisch schonenden Vorgehen. Damit wird diese Technologie zugänglicher für Forschungslabore weltweit.Zusammenfassend steht TEMI für eine innovative Kombination von Gewebeexpansion und Massenspektrometrie-Bildgebung, die eine hochauflösende, umfassende und multimodale molekulare Kartierung in Gewebeproben ermöglicht. Diese Methode adressiert zentrale Limitationen konventioneller Ansätze, eröffnet neue Einblicke in die molekulare Organisation gesunder und krankhafter Gewebe und ist vielversprechend für die Weiterentwicklung von Biowissenschaften und Medizin.