Die Frage, wie Moleküle zu Zeichen wurden, beschäftigt nicht nur Biologen, sondern auch Philosophen, Informationswissenschaftler und Forscher aus dem Bereich der Biosemiotik. Hinter dieser scheinbar einfachen Frage verbirgt sich ein komplexes Zusammenspiel aus Physik, Chemie, Evolution und Interpretation. Die Entwicklung von Molekülen, die nicht nur chemische Objekte sind, sondern als Träger von Information fungieren, markiert einen entscheidenden Schritt in der Entstehung des Lebens und des biologischen Verständnisses von Zeichen und Bedeutung. Im Zentrum dieser Diskussion steht die Herausforderung, zu verstehen, wie physikalische Moleküle wie DNA oder RNA ihre Funktion als Informationsübermittler gewinnen konnten. Klassische molekularbiologische Theorien betonen DNA und RNA als Ursprung und Träger biologischer Information.
Dennoch wirft eine genauere Analyse Zweifel an der reinen Reproduktion dieser Information auf, da reine Kopierprozesse keine intrinsische Bedeutung oder Funktion implizieren. Die spannende Alternative, die sich daraus ergibt, besteht darin, dass die biologischen Moleküle nicht selbst die primären Informationsträger sind, sondern semiotische Artefakte, welche die interpretativen Fähigkeiten eines Systems nutzen. Die Biosemiotik, als interdisziplinäres Forschungsgebiet, untersucht genau dieses Phänomen. Sie betrachtet semiosis als den Prozess, durch den Bedeutungen erzeugt und interpretiert werden. Für Moleküle bedeutet das, dass gewisse molekulare Eigenschaften selbst nicht automatisch Bedeutung tragen, sondern dass erst ein interpretierendes System diese Eigenschaften als Zeichen erkennt und sinnvoll nutzt.
Dabei ist der Fokus auf das interpretierende System essenziell, denn erst dieses verleiht den molekularen Strukturen ihre semantische Relevanz. Historisch wurde das Problem des Lebens und der biologischen Information bereits von Erwin Schrödinger angegangen, der 1944 in seinem Werk „Was ist Leben?“ konstatierte, dass lebende Organismen trotz thermodynamischer Unordnung beständig Ordnung und Organisation aufrechterhalten. Seine Vorstellung von einem „aperiodischen Kristall“ als Träger genetischer Information wurde in den 1950er Jahren durch die Entdeckung der DNA-Doppelhelixstruktur von Watson und Crick wissenschaftlich untermauert. Im Anschluss daran verfestigte sich das Dogma der molekularbiologischen Informationsweitergabe: DNA zu RNA zu Protein. Doch dieses Dogma enthielt eine Reduktion von Information auf ein rein physikalisches Muster und deren exakte Replikation.
Die Einsicht, dass Replikation allein keine Bedeutung hervorbringt, unterstreicht die Notwendigkeit eines interpretativen Prozesses für biologische Information. Die Unterscheidung zwischen reiner Datenübermittlung und Bedeutung – also zwischen Shannon’s mathematischer Informationstheorie und semantischer Information – wurde von Informationstheoretikern wie Robert Fano betont. Die biologische Realität verlangt, dass molekulare Muster im Kontext ihrer Funktion und Bedeutung innerhalb des lebenden Systems interpretiert werden. Um zu verstehen, wie solche Interpretationsprozesse auf molekularer Ebene entstehen können, ist die Entwicklung von minimal komplexen Modellsystemen entscheidend. Eines der zentralen Modelle ist das der Autogenese: ein hypothetisches, aber physikalisch realisierbares molekulares System, das selbständig Replikation und Selbstreparatur betreibt, basierend auf reziproker Katalyse und Selbstassemblierung.
Die Autogenese verbindet zwei fundamentale molekulare Prozesse – reziproke Katalyse, bei der Produkte einer Reaktion als Katalysatoren für eine andere Reaktion fungieren, und die Fähigkeit bestimmter Moleküle, sich spontan zu einer strukturierten Kapsel zu assemblieren. Die Kapsel schützt dabei die katalytischen Bestandteile und hält diese lokalisiert, was die Wahrscheinlichkeit ihrer Interaktion erhöht. Dieses Zusammenspiel erzeugt eine dynamische Wechselwirkung, in der sich das System selbst wiedererzeugt und repariert. Es handelt sich damit um einen minimalen autogenen Arbeitszyklus, vergleichbar mit einem Motor, der sich durch Energiezufuhr über einen Zyklus hinweg selbst erneuert. Die Bedeutung dieses Modells liegt in seiner Interpretation als erstes Zeichen- und Informationssystem auf molekularer Ebene.
In einem autogenen Zyklus wird jede Form von Störung als Signal (ein Zeichen) für Nicht-Selbst erkannt, das durch den Reparaturprozess respondiert wird. Dabei handelt es sich um eine Form der ikonischen Interpretation, in der das Zeichen direkt durch Ähnlichkeit mit dem zu erkennenden Zustand verbunden ist – im Unterschied zu komplexeren semiotischen Prozessen mit Indexen oder Symbolen. Steigt die Komplexität eines solchen Systems leicht an, etwa durch die Fähigkeit, Umgebungsbedingungen zu detektieren und in die Dynamik einzubeziehen, entsteht eine Form von indexikalem Zeichenverstehen. Zum Beispiel könnte die Kapsel eines Autogens Oberflächenmerkmale besitzen, an die Substratmoleküle binden. Diese Bindung erhöht die Bruchwahrscheinlichkeit der Kapsel, was wiederum die Freisetzung der katalytischen Moleküle in günstiger Umgebung induziert.
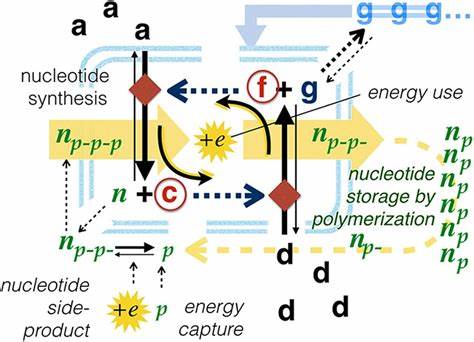
Solch eine Fähigkeit erlaubt dem System eine adaptive Reaktion auf Umweltzeichen, die seine Selbstreproduktion begünstigen – eine wesentliche Voraussetzung für evolutionäre Dynamik. Der nächste evolutionäre Schritt ist die Verschiebung der Informationsspeicherung auf lineare Nukleotidpolymere wie RNA oder DNA. Ursprünglich dürften diese Polymere energetische Rollen erfüllt haben, wie Freeman Dyson argumentierte, indem sie energiereiche Phosphatbindungen trugen und so als Energiespeicher fungierten. Später wurden diese Moleküle zu Informationsspeichern umgedeutet – einer sogenannten Exaptation, bei der ein bereits existierendes Element eine neue Funktion übernimmt. Die Polymerstruktur von Nukleotiden erlaubt eine relativ stabile Speicherung von „Beschränkungen“ (constraints), die nicht direkt energetischen, sondern informativen Zwecken dienen.
Die Sequenz von Nukleotiden beeinflusst die dreidimensionale Struktur des Moleküls, was wiederum deren Bindung und Interaktion mit anderen Molekülen steuert. Diese physikalische Grundlage schafft eine semiotische Brücke zwischen der Information im Molekül und seiner funktionalen Umsetzung – ein wesentlicher Schritt hin zur echten biologischen Bedeutung. Durch diese Vermittlung entsteht ein Code-ähnliches System, das über die einfache Ähnlichkeit hinausgeht. Es handelt sich um eine arbiträre, aber stabile Beziehung zwischen zwei völlig unterschiedlichen molekularen Substraten, die durch adaptierende Elemente wie tRNA-Moleküle gekoppelt werden. Genau hier zeigt sich der evolutionäre Sprung: Moleküle werden Zeichen, weil ihre strukturellen Eigenschaften korreliert und gekoppelt werden mit der Funktion bestimmter Reaktionen in einem interpretierenden System.
Das Verschieben von funktionalen Beschränkungen von dynamischen Katalysatoren auf statische template Moleküle ermöglicht ein sich selbst rekurrierendes Informationssystem, das sich iterativ weiterentwickeln kann. Diese Struktur bildet die Grundlage für die hohe Evolvabilität der biologischen Systeme und für eine zunehmende Komplexität der Interpretation. Das Konzept der semiotischen Gerüste („semiotic scaffolding“) fasst diese Hierarchie interpretativer Ebenen zusammen. Es beschreibt, wie aufeinander aufbauende interpretierende Prozesse auf verschiedenen molekularen und zellulären Ebenen komplexere Informationsverarbeitung und Regulation ermöglichen. Beispielsweise regulieren DNA-Sequenzen die Struktur von Proteinen, welche wiederum Genexpression kontrollieren können.
So entsteht ein selbstorganisierendes System von Informationsweitergabe und Interpretation, das sich auf höheren Ebenen in Geweben, Organismen und sogar sozialen Systemen fortsetzt. Diese Perspektive stellt das klassische Bild von DNA und RNA als alleinigem Ursprung biologischer Information auf den Kopf. Stattdessen erscheinen diese Moleküle als semiotische Artefakte, die erst durch ihre Einbettung in interpretierende molekulare Systeme zu Informationsträgern werden. Das betont den entscheidenden Stellenwert der Interpretation und der dynamischen Beziehungen, die semantische Bedeutung erzeugen. Zusammenfassend zeigt die Analyse, dass Moleküle erst durch spezielle interpretative Prozesse und dynamische Kopplungen zu Zeichen werden, die biologische Information enthalten und weitergeben.
Die Autogenese liefert einen minimalen, chemisch plausiblen Mechanismus für die Entstehung erster semiotischer Systeme. Der evolutionäre Übergang zur Nutzung von Nukleotidpolymeren als Template bietet die Basis für die genetische Informationsspeicherung, während die hierarchische semiotische Gerüstbildung eine Erklärung für die enorme Komplexität biologischer Informationsverarbeitung darstellt. Das Verständnis, wie Moleküle zu Zeichen werden, hat nicht nur fundamentale theoretische Bedeutung, sondern auch Implikationen für die Erforschung des Ursprungs des Lebens, synthetische Biologie und die Entwicklung bio-inspirierter Informationssysteme. Indem Forschung an der Schnittstelle von Chemie, Biologie und Semiotik betrieben wird, eröffnen sich neue Wege, das Wesen von Information und Bedeutung in natürlichen Systemen begreifen zu lernen – eines der großen ungelösten Rätsel der Wissenschaft.




![Why Prescription Drug Prices in the US Are So High [2024]](/images/CA9C4C95-F9D9-4F5A-AA4E-1E7795375E4D)



