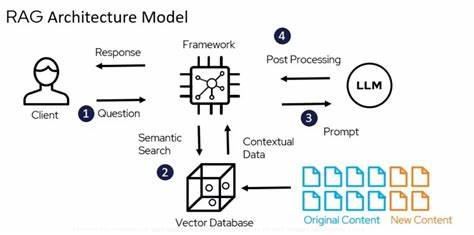
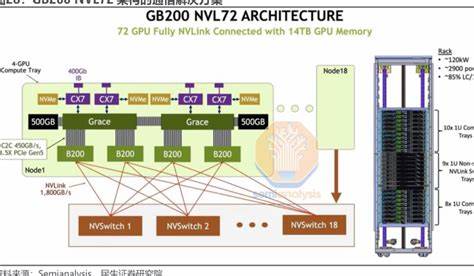
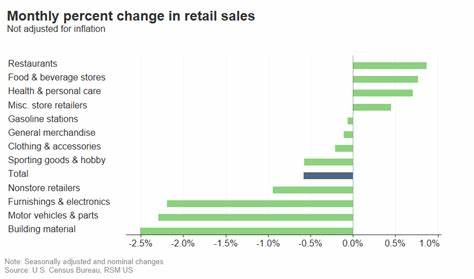
Die Wissenschaft der Proteine ist ein zentrales Element der modernen Biotechnologie und Medizin. Ihre Bedeutung erstreckt sich von der Grundlagenforschung bis zu Anwendungen wie der Medikamentenentwicklung und personalisierter Medizin. Mit dem Fortschritt der künstlichen Intelligenz ist das Verständnis von Proteinen auf textlicher Basis zu einem wegweisenden Forschungsfeld avanciert. Dabei stellt sich die Frage, ob klassische Retrieval-Methoden oder Large Language Models (LLMs) die bessere Wahl für die Analyse und Generierung von Proteintexten sind. Diese Diskussion ist zentral für die weitere Evolution von Technologien, die die Proteinforschung beschleunigen und präzisieren sollen.
Protein-Text-Modelle haben in den letzten Jahren erheblich an Bedeutung gewonnen. Diese Modelle ermöglichen es, Proteindaten nicht nur als Sequenzen chemischer Bausteine zu betrachten, sondern auch in einem sprachbasierten Kontext zu verstehen. Das Ziel ist es, biologisches Fachwissen und Textinformationen miteinander zu verknüpfen, sodass komplexe biologische Zusammenhänge besser interpretiert und genutzt werden können. Durch die Integration von Proteininformationen in LLMs sollen zum Beispiel Zusammenhänge zwischen Struktur, Funktion und genetischen Variationen effizienter erkannt und erklärt werden. Traditionell wurden Retrieval-Methoden eingesetzt, bei denen große Datenbanken nach relevanten Informationen durchsucht werden.
Diese Vorgehensweise beruht auf der Annahme, dass das benötigte Wissen in bestehenden Datensätzen vorhanden ist und lediglich gefunden werden muss. Bei der Proteinforschung bedeutet das oft, dass Sequenzen, experimentelle Befunde oder wissenschaftliche Publikationen durchsucht werden, um Antworten auf Fragestellungen zu finden. Retrieval-Systeme zeichnen sich durch ihre Effizienz und Zuverlässigkeit aus, besonders wenn es darum geht, präzise Informationen in großen Textmengen zu identifizieren. Mit dem Aufkommen von LLMs, die auf Transformer-Architekturen basieren, änderte sich die Dynamik grundlegend. Diese Modelle werden mit enormen Textmengen trainiert und können anschließend kontextualisierte, kreative und umfangreiche Antworten generieren.
Im Bereich der Proteinforschung versprechen sie, komplexe Zusammenhänge aus der Literatur zu erschließen, Hypothesen zu generieren und sogar potenzielle Proteinstrukturen oder Funktionen basierend auf textuellen Daten vorherzusagen. Die Flexibilität und Anpassungsfähigkeit von LLMs machen sie zu einem mächtigen Werkzeug in der bioinformatischen Analyse. Ein kritisches Problem, das in der jüngsten Forschung hervorgehoben wurde, ist jedoch die Datenlecksituation bei bestehenden Benchmarks für textbasiertes Proteinverständnis. Viele der verwendeten Datensätze enthalten nicht nur Trainingsdaten, sondern auch Testdaten, die zuvor zugänglich waren. Das führt zu einer Überschätzung der Leistungsfähigkeit von Modellen, da sie Informationen aus dem Training bereits kennen und somit auf Testfragen scheinbar präzise antworten können.
Dies wirkt sich negativ auf die Validität und Verlässlichkeit der bewerteten Modelle aus und verzerrt die Vergleichbarkeit verschiedener Methoden. Darüber hinaus sind herkömmliche Metriken, die ursprünglich für die Bewertung von natürlichsprachlichen Modellen entwickelt wurden, für die Proteinanalyse nur begrenzt geeignet. Worte und Satzstrukturen in der Biologie haben eine andere Bedeutung und Komplexität als im allgemeinen Sprachgebrauch. Die Bedeutung biologischer Entitäten, etwa Protein-Domänen, Aminosäuresequenzen oder funktionelle Motive, können durch Standardmetrikverfahren nicht adäquat erfasst werden. Dies führte zur Entwicklung eines neuen Evaluationsrahmens, der spezifisch auf biologische Entitäten ausgerichtet ist und eine realistischere Beurteilung der Modellleistung ermöglicht.
Vor diesem Hintergrund haben Forscher neue Datensätze reorganisiert und diese innovative Bewertungsmethodik etabliert. Dies ist ein wesentlicher Schritt, um zukünftige Entwicklungen auf eine solide Basis zu stellen und Vergleiche zwischen LLM-basierten und Retrieval-basierten Ansätzen fair und transparent zu machen. Die Ergebnisse zeigen, dass eine retrievalbasierte, also suchgestützte Methode, welche relevante Proteindaten effizient filtert und einbindet, in vielen Fällen die Leistung von feinjustierten LLMs übertrifft. Besonders hervorzuheben ist die Effizienz und Genauigkeit eines retrievalverstärkten Verfahrens, das ohne aufwendiges Training oder Feinabstimmung auskommt. Dies eröffnet spannende Perspektiven für Szenarien, in denen Ressourcen knapp sind oder schnelle Resultate benötigt werden.
Im Vergleich dazu erfordern LLM-Ansätze häufig umfangreiche Rechenleistung und Trainingsdaten, was ihre praktische Einsetzbarkeit einschränken kann. Der Einsatz von Retrieval-Systemen ergänzt die Möglichkeiten der LLMs und kann in vielen Fällen zu einer Verbesserung der Textgenerierung von Proteininformationen führen. Durch die Kombination beider Ansätze entsteht ein hybrides System, das sowohl auf dem reichen Wissen großer Datenbanken als auch auf der adaptiven Fähigkeit der LLMs basiert, komplexe Zusammenhänge zu modellieren. Dieses Zusammenspiel könnte den Weg für innovative Werkzeuge ebnen, die die Proteinforschung schneller, genauer und zugänglicher machen. In Zukunft ist zu erwarten, dass die Integration von biologischem Fachwissen, verbesserten Bewertungsmethoden und fortschrittlichen KI-Technologien wie LLMs und Retrieval-Systemen die biomedizinische Forschung revolutionieren wird.
Die Herausforderungen in der Bewertung und die Probleme der Datenlecks zeigen aber, dass eine sorgfältige Methodik und realistische Tests essenziell sind, um wirklich brauchbare und robuste Modelle zu entwickeln. Insgesamt ist die Diskussion um Retrieval versus Large Language Models im Bereich des textbasierten Proteinverständnisses ein entscheidender Impulsgeber für die bioinformatische Community. Es unterstreicht die Notwendigkeit, innovative Ansätze kritisch zu hinterfragen und die Forschung auf nachhaltige und validierbare Methoden zu konzentrieren. Nur so kann das volle Potenzial der Künstlichen Intelligenz für das Verständnis der komplexen Welt der Proteine ausgeschöpft werden.