Die Erforschung der dreidimensionalen Genomarchitektur hat in den letzten Jahren neue Horizonte in der Krebsforschung eröffnet. Im Zentrum steht die Erkenntnis, dass das menschliche Genom nicht nur linear existiert, sondern im Zellkern komplex gefaltet und räumlich organisiert ist. Diese 3D-Struktur beeinflusst maßgeblich, wie Gene reguliert werden und wie sich Tumore entwickeln. Die jüngste Studie zur dreidimensionalen Genomlandschaft primärer menschlicher Krebsgeschwülste liefert bislang unvergleichliche Einblicke in diese Mechanismen und zeigt auf, wie differenzierte Genregulation bei verschiedenen Krebsarten erfolgt. In der Zelle stellt sich die etwa zwei Meter lange DNA als fein gefaltetes Molekül im winzigen Zellkern von ungefähr 10 Mikrometern Durchmesser dar.
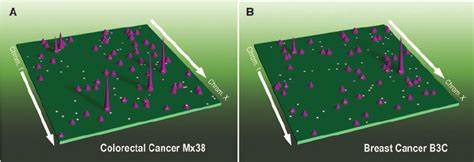
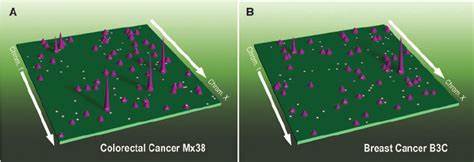
Die dreidimensionale Organisation erlaubt es regulatorischen Elementen, sogenannten Enhancern, mit Genen auch über große chromosomale Distanzen dynamisch zu interagieren, wodurch eine präzise und zelltypspezifische Genexpression ermöglicht wird. Diese Vorgänge beruhen auf der komplexen Strukturierung des Genoms in sogenannte A- und B-Kompartimente, die aktive (euchromatische) und inaktive (heterochromatische) Bereiche trennen, sowie auf Topologisch Assoziierten Domänen (TADs), welche als räumliche Einheiten innerhalb von Chromosomen dienen und intermolekulare Kontakte begrenzen. Die Studie analysierte 69 Tumorproben aus 15 verschiedenen menschlichen Primärtumoren, darunter Brust-, Leber-, Darm- und Lungenkrebs, unter Verwendung einer Methode namens H3K27ac HiChIP. Diese Technik ermöglicht es, Protein-gebundene Genominteraktionen mit einer Markierung für aktive Enhancer simultan zu erfassen. Die Kombination dieser Daten mit tiefgehender Ganzgenomsequenzierung (WGS) und Einzelzell-Erreichbarkeitsanalysen bietet eine multidimensionale Perspektive auf das Zusammenspiel von Genomstruktur, Genregulation und Krebsentwicklung.
Eines der zentralen Ergebnisse ist die Beschreibung von drei Archetypen der Enhancer-Nutzung, die über 100 Onkogene in den verschiedenen Krebsarten charakterisieren. Einige Gene zeigen eine statische Enhancerbindung, die über verschiedene Tumorarten hinweg erhalten bleibt. Andere Onkogene hingegen weisen spezifisch in bestimmten Tumoren eine gezielte Hinzunahme neuer Enhancerkontakte auf. Wieder andere zeigen ein dynamisches Muster des Enhancer-Rewiring mit weitreichenden Veränderungen in der Vernetzung, was auf eine flexible Genregulationslandschaft in Tumoren hindeutet. Ein weiters Augenmerk gilt der Rolle von strukturellen Variationen (SVs) im Krebsgenom.
Krebsgeschwüre sind bekannt für ihre genomische Instabilität, die beispielsweise durch Deletionen, Duplikationen, Inversionen und Translokationen charakterisiert ist. Diese SVs können die 3D-Architektur des Genoms stark verändern und neue Interaktionen zwischen Enhancern und Promotoren schaffen, was wiederum zur Aktivierung von Onkogenen führt. Besonders bemerkenswert sind extrachromosomale DNA-Elemente (ecDNA), die als kreisförmige DNA-Moleküle vorliegen, massenhaft Onkogene amplifizieren und mit aggressiveren Krankheitsverläufen korrelieren. Die Studie weist darauf hin, dass ecDNA-assoziierte Amplifikationen die umfangreichsten Wechselwirkungen und Rekonfigurationen in der Enhancer-Vernetzung hervorrufen, was ihre prominente Rolle bei der Tumorprogression weiter unterstreicht. Die dreidimensionale Genomanalyse ermöglichte außerdem die präzise Identifikation von Allel-spezifischen Mutationen in nichtkodierenden Regionen, die eine erhöhte Enhancer-Aktivität und gezieltes Rewiring der Genregulation bewirken.
So konnten bestimmte nichtkodierende Punktmutationen als Mechanismen entdeckt werden, die über die Bildung neuer Transkriptionsfaktor-Bindungsstellen zur Überexpression von Onkogenen beitragen. Diese Erkenntnisse eröffnen neue Wege in Richtung personalisierter Medizin, indem sie nicht nur Mutationen in Genen selbst, sondern auch deren regulatorische „Steuerungselemente“ in den Blick rücken. Neben dem Tumor selbst rückt auch das Tumormikromilieu (TME) in den Fokus. Das TME besteht aus einer komplexen Gemeinschaft von verschiedenen Zelltypen, darunter Immunzellen verschiedenster Art, die aktiv die Tumorentwicklung beeinflussen. Die Integration von HiChIP-Daten mit Einzelzellchromatin-Erreichbarkeitsdaten (scATAC-seq) ermöglichte die Zuordnung von Enhancer-Promoter-Wechselwirkungen zu spezifischen Zelltypen innerhalb des Tumorgewebes.
So konnte beispielsweise gezeigt werden, dass wichtige Immun-Checkpoints wie PD-L1 (CD274) von Enhancern reguliert werden, die spezifisch in myeloiden Immunzellen aktiv sind. Diese Zellentyp-spezifische Genregulation bestätigt die Bedeutung der Immunzellen im TME für die Immunflucht von Tumoren und die Antwort auf Immuntherapien. Die differenzierte Betrachtung der 3D-Genomstruktur bringt zudem zutage, dass niedrigere Ordnungsebenen wie Kompartimente und TADs bei verschiedenen Krebsarten vergleichsweise stabil sind. Größere Unterschiede manifestieren sich vor allem in den feinkörnigen Enhancer-Promoter-Schleifen, die als maßgebliche Treiber der Tumortyp-spezifischen Genexpression gelten. Diese subtilen Anpassungen der Genomtopologie leisten einen wesentlichen Beitrag zur Diversität der Krebsbiologie und eröffnen Ansatzpunkte für die gezielte Modulation.
Im klinischen Kontext resultieren diese neuen Einblicke in der 3D-Genomlandschaft in vielversprechenden diagnostischen und therapeutischen Perspektiven. Erstens kann die Analyse von Kopienzahlveränderungen und Enhancer-Rewiring zusammen dazu beitragen, die Expression von Onkogenen besser vorauszusagen und potenzielle Biomarker für prognostische oder therapeutische Entscheidungen zu identifizieren. Zweitens legt die Definition von Zelltyp-spezifischen Regulome im TME eine personalisierte Immuntherapie nahe, welche gezielt auf die Regulation von Immun-Checkpoints in bestimmten Immunzelltypen eingeht. Die Forschungsarbeiten zeigen auch, dass die Evolution der Tumorzellen durch genomische Instabilität auf mehreren Ebenen vorangetrieben wird. Komplexe Strukturrearrangements führen zu neuartigen Enhancer-Promoter-Kontakten, gefördert durch die Mobilität von ecDNA, die nicht an chromosomale Topologisch Assoziierte Domänen gebunden ist.
Dies ermöglicht eine schnelle genetische Anpassung und Diversifizierung, die für das Therapieversagen und die Metastasierung von Krebserkrankungen von hoher Bedeutung sein kann. Insgesamt unterstreicht die dreidimensionale Analyse des Krebsgenoms die Notwendigkeit, über eine rein lineare Genomdarstellung hinauszudenken. Das Genom ist ein dynamisches Fluktuieren räumlicher Wechselwirkungen, das entscheidend für die Aktivierung oder Repression von Genen ist. Besonders in der Krebsentwicklung zeigen sich diese Mechanismen als Triebfedern der Tumorprogression und Heterogenität. Die Integration multidimensionaler Daten – von epigenetischen Markern über Genomsequenzen bis hin zu Einzelzellanalysen – liefert ein bisher nicht erreichtes Verständnis der molekularen Landschaft in Krebs.
Langfristig verspricht die Einbindung dreidimensionaler Genomanalytik neue diagnostische Biomarker und innovative Therapieansätze, zum Beispiel durch gezielte Störung pathologischer Enhancer-Promoter-Schleifen oder durch das Ausschalten kritischer ecDNA-Strukturen. Durch das bessere Verständnis der komplexen Regulierung von Onkogenen und der Interaktionen im Tumormikromilieu könnten personalisierte Therapien optimiert und resistenzfördernde Mechanismen umgangen werden. Die aktuellen Ergebnisse markieren daher einen zentralen Fortschritt in der Krebsforschung, der die Grundlagen für die nächste Generation molekularer Diagnostik und Therapien legen kann. Während viele Fragen noch offen sind, schafft die Karte der dreidimensionalen Genomlandschaft eine neue Perspektive, um die Komplexität von Krebs auf systemischer Ebene zu verstehen und gezielt therapeutisch anzugreifen.