Die Erforschung der dreidimensionalen Genomlandschaft primärer menschlicher Krebserkrankungen hat in den letzten Jahren bedeutende Fortschritte erzielt und revolutioniert unser Verständnis von Krebsentstehung und -entwicklung. Die traditionelle Sicht auf das menschliche Genom, verstanden als lineare Abfolge von DNA-Basen, wird zunehmend durch das Wissen über seine räumliche Organisation im Zellkern ergänzt. Diese komplexe dreidimensionale Faltung spielt eine entscheidende Rolle für die Regulation der Genexpression, die Zellfunktion und letztlich auch für die Pathogenese von Krebs. In jeder menschlichen Zelle ist die genomische DNA, bestehend aus etwa zwei Metern an Molekülsträngen, kompakt in einem winzigen Zellkern mit einem Durchmesser von ungefähr zehn Mikrometern untergebracht. Das Genom ist nicht zufällig gefaltet, sondern weist eine hierarchische Strukturierung auf, die in verschiedenen Skalen funktioniert.
Chromosomen sind in sogenannte A- und B-Kompartimente unterteilt, welche aktive bzw. inaktive Genbereiche repräsentieren. Zusätzlich sind Topologisch Assoziierte Domänen (TADs) wichtige regulatorische Domänen, die Gene und ihre distalen Regulatoren innerhalb der gleichen Domäne zusammenfassen und die Interaktion über Domänengrenzen hinweg erschweren. Der bedeutendste Aspekt dieser Organisation für die Krebsforschung ist die Rolle der Enhancer, die als distal gelegene regulatorische DNA-Elemente mit Promotoren von Zielgenen durch Schleifen oder dreidimensionale Kontaktstellen verbunden sind. Diese Enhancer-Promotor-Interaktionen ermöglichen eine präzise und zelltypspezifische Genregulation.
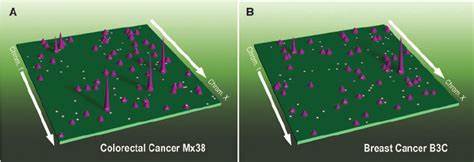
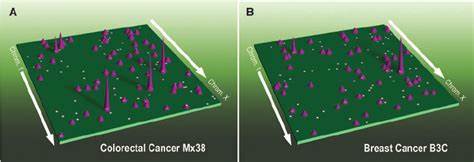
Veränderungen in der dreidimensionalen Organisation des Genoms können folglich zu Fehlregulationen von Genen führen, insbesondere von solchen, die maßgeblich an der Krebsentstehung beteiligt sind, wie Onkogene. Die jüngste groß angelegte Studie, die 69 Tumorproben aus 15 verschiedenen primären menschlichen Krebsarten analysierte, liefert bahnbrechende Erkenntnisse zur dreidimensionalen Genomstruktur in Tumorzellen. Mittels einer Methode namens HiChIP, die Protein-vermittelte Chromosomenfaltung und gleichzeitige Aktivitätsmessung von Enhancern durch H3K27ac-histone-Acetylierung kombiniert, wurde ein detailliertes Bild der Enhancer-Promotor-Verbindungen gewonnen. Diese Analyse führte zur Identifikation von über 665.000 signifikanten chromatinbasierten Interaktionen, von denen ein erheblicher Teil neuartig ist und zuvor in anderen Datensätzen nicht erfasst wurde.
Ein zentrales Ergebnis dieser Forschung ist die Klassifikation von über 100 Onkogenen hinsichtlich ihrer Enhancer-Nutzungsmuster in verschiedenen Krebsarten. Es lassen sich drei grundlegende Archetypen bestimmen: statische Nutzung, selektiver Gewinn neuer Enhancer-Verbindungen und dynamisches Rewiring, also eine Neuausrichtung der Enhancer-Promotor-Interaktionen. Dieses heterogene Verhalten verdeutlicht, wie unterschiedlich Krebszellen verschiedene regulatorische Mechanismen verwenden können, um Onkogene zu aktivieren. Darüber hinaus ermöglicht die Kombination aus tiefer Ganzgenomsequenzierung und 3D-Genomkartierung eine präzise Aufdeckung struktureller Variationen (SVs), wie zum Beispiel Genomduplikationen, Translokationen, Inversionen und amplifizierte Genregionen. Diese komplexen genomischen Veränderungen beeinflussen direkt die dreidimensionale Organisation und können neue Interaktionslandschaften schaffen, die etwa regulatorische Elemente an Onkogene heranführen und deren Überexpression fördern.
Besonders hervorzuheben ist die Rolle von extrachromosomaler DNA (ecDNA), die als kreisförmiges DNA-Molekül im Zellkern vorkommt und eine intensive Verstärkung von Onkogenen ermöglicht. Diese ecDNA ist verantwortlich für ein deutlich ausgeprägtes Rewiring von Enhancer-Netzwerken und wird mit einer schlechten Prognose in Verbindung gebracht. Neben den genetischen und epigenetischen Veränderungen in den eigentlichen Krebszellen spielt auch das Tumormikromilieu eine wichtige Rolle. Die Studie zeigt, dass das 3D-Genom differenziert betrachtet werden muss, um sowohl die Malignität der Tumorzellen als auch die Aktivität im umgebenden Immunsystem zu verstehen. Durch die Integration von Einzelzell-ATAC-seq-Daten, welche chromatinzugängliche Regionen in einzelnen Zelltypen des Tumors messen, konnten zelltypspezifische Enhancer-Promotor-Interaktionen im Tumormikromilieu aufgelöst werden.
Hierbei wurde unter anderem eine myeloidzellspezifische Regulation des Immun-Checkpoint-Gens CD274 (PD-L1) identifiziert, was die wichtige Rolle tumorassoziierter Makrophagen und anderer Immunzellen betont. Die dreidimensionale Genomforschung liefert zudem wertvolle Einsichten hinsichtlich nichtkodierender Mutationen, die bisher meist schwer zu interpretieren waren. Die Kombination von somatischen Mutationsdaten, HiChIP-assoziierter Verstärkung der H3K27ac-Markierung und 3D-Interaktionsprofilen ermöglicht es, Mutationen zu erkennen, die funktional neue Transkriptionsfaktor-Bindungsstellen erzeugen und damit die Aktivierung von Enhancern und Onkogenen in einem patientenspezifischen Kontext bewirken. Solche Erkenntnisse eröffnen Perspektiven für präzise diagnostische Instrumente und gezielte Therapieoptionen. Die Erkenntnisse aus der Analyse der dreidimensionalen Genomstruktur in Krebs sind auch für die Weiterentwicklung personalisierter Medizin von großer Bedeutung.
Das Verständnis, wie bestimmte Onkogene durch CNV (Kopienzahlvariationen) oder durch Enhancer-Aktivität reguliert werden, hilft bei der Identifikation von Patienten, die für bestimmte zielgerichtete Therapien in Frage kommen. Zudem kann die Charakterisierung der etablierten chromosomalen Neuanordnungen und deren Auswirkungen auf die Genregulation neue therapeutische Angriffspunkte darstellen. Trotz dieser enormen Fortschritte bleiben Herausforderungen bestehen. Die Heterogenität der Krebsarten, die interindividuelle Variabilität und die komplexen Wechselwirkungen im Tumormikromilieu erfordern eine weitere Vertiefung der Analysen. Auch die technischen Limitationen hinsichtlich der Sequenzierungstiefe und der Auflösung von 3D-Genomkartierungen müssen überwunden werden, um das volle Potenzial dieser Erkenntnisse im klinischen Alltag zu entfalten.
Insgesamt veranschaulicht die dreidimensionale Genomanalyse von primären menschlichen Krebserkrankungen die Dynamik und Plastizität des tumorassoziierten Genoms in einem neuen Licht. Sie hebt die zentrale Rolle der räumlichen Organisation des Erbguts als Regulator von Genexpression hervor und unterstreicht die Bedeutung der integrierten Betrachtung von genetischen, epigenetischen und zellulären Kontexten im Kampf gegen Krebs. Die gewonnenen Daten und Erkenntnisse bilden eine wertvolle Ressource, die zukünftige Forschungsprojekte antreiben und die Entwicklung innovativer diagnostischer und therapeutischer Strategien ermöglichen werden.