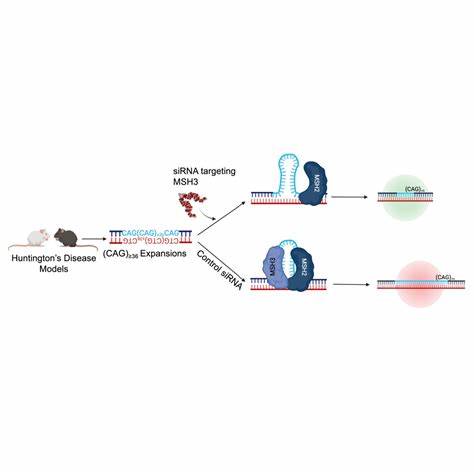
Die Huntington-Krankheit (HD) ist eine verheerende neurodegenerative Erkrankung, die genetisch durch die Expansion von trinukleotidischen Wiederholungen, insbesondere der CAG-Sequenz im HTT-Gen, verursacht wird. Diese Wiederholungen kodieren für Polyglutamin (PolyQ)-Abschnitte im Huntingtin-Protein, deren Länge entscheidend für den Krankheitsbeginn, den Verlauf und die Schwere ist. Längere CAG-Repeat-Trakte führen häufig zu früherem Ausbruch und schwereren Symptomen. Ähnlich verhält es sich bei anderen Erkrankungen, wie etwa Friedreich-Ataxie (FRDA), die auf die Expansion von GAA-Wiederholungen im FXN-Gen zurückzuführen ist. Die Herausforderung bei solchen Erkrankungen liegt darin, dass die so genannten trinukleotidische Wiederholungen (TNR) im Körper somatisch instabil sind und sich im Laufe des Lebens weiter ausdehnen können, was den Krankheitsverlauf verschärft.
Eine erfolgversprechende Strategie, um diesen Teufelskreis zu durchbrechen, ist die Nutzung moderner Genome Editing-Technologien, insbesondere des Base Editing, um gezielt Veränderungen in den Repeat-Trakten herbeizuführen. Base Editing ist eine innovative Form der genetischen Manipulation, bei der gezielt einzelne Nukleotidbasen in der DNA ausgetauscht werden, ohne dass dabei DNA-Doppelstrangbrüche entstehen, wie sie bei CRISPR/Cas9 mit herkömmlicher Schneidetechnik vorkommen. Speziell für Huntington und FRDA wurden cytosinbasierte (CBE) und adeninbasierte Base Editoren (ABE) verwendet, um gezielte Veränderungen innerhalb der pathogenen Repeat-Sequenzen anzubringen. Ziel dabei ist es, innerhalb der langen Wiederholungsstrecken einzelne Nukleotide so zu verändern, dass natürliche, meist stabile Unterbrechungen simuliert werden. Solche Interruptions werden in Populationen gefunden und sind mit weniger somatischer Instabilität, milderen Symptomen und verzögertem Krankheitsbeginn assoziiert.
Forscher haben gezeigt, dass das Einbringen von CAA-Unterbrechungen in die CAG-Wiederholungen des HTT-Gens die anfängliche Längenzunahme der Repeats in patienteneigenen Zellen deutlich reduziert. Diese Veränderungen erfolgen synonym, das heißt, sie verändern nicht die kodierte Aminosäure – Glutamin bleibt erhalten – und somit bleibt die Proteinfunktion erhalten, während der genomische Repeat stabilisiert wird. Die Methode beruht darauf, die cytosinbasierte Base Editing-Technologie gezielt auf die CTG-Sequenzen, die der CAG-Sequenz am Gegenstrang entsprechen, anzuwenden. Das Ergebnis sind stabile, nicht-pathogene Allele, die den Fortschritt der Repeat-Expansion dämpfen. In präklinischen Modellen, insbesondere in genetisch modifizierten Mäusen mit langen HTT-CAG-Repeats (Htt.
Q111), konnte diese Technologie mittels AAV9-Vektor erfolgreich in das zentrale Nervensystem (CNS) eingebracht werden. Die Tiere erhielten die Base Editoren direkt nach der Geburt über intraventrikuläre Injektionen. Die Studien zeigten, dass ein signifikanter Anteil der Pathogen-Allele mit den gewünschten CAA-Unterbrechungen versehen wurde, was mit einer signifikanten Verringerung der somatischen Expansion der CAG-Trakte im Cortex und Striatum einherging. Damit wurde erstmals eine Methode demonstriert, die nicht nur die Expansion verhindert, sondern auch bestehende Repeat-Trakte verkürzen kann, was besonders wichtig für Patienten mit bereits langer CAG-Repeats ist. Parallel wurde die adeninenbasierte Base Editing-Technologie (ABE) eingesetzt, um die GAA-Repeat-Trakte im FXN-Gen bei Friedreich-Ataxie zu verändern.
Dabei wurden Interruptions in Form von GAG oder GGA Triplets eingeführt, die ebenfalls natürlich vorkommen und mit milderen Krankheitsverläufen in Verbindung gebracht werden. In patienteneigenen Fibroblasten konnte die Transkription von FXN verbessert und die Repeat-Expansion signifikant reduziert werden. In den entsprechenden Mausmodellen (YG8s) wurde eine vergleichbare Reduktion der somatischen GAA-Expansionen im Cortex durch AAV9-vermittelte ABE-Expression erreicht. Dies unterstreicht das breite Anwendungspotenzial des Base Editing bei verschiedenen TNR-bedingten Erkrankungen. Ein bedeutsamer Vorteil dieser base editingbasierten Intervention liegt in ihrer Präzision und der im Vergleich zu klassischen Genom-Editing-Ansätzen viel geringeren Anzahl unerwünschter DNA-Doppelstrangbrüche.
Diese Minimierung von DNA-Schäden reduziert potenzielle unerwünschte Mutationen oder zelluläre Stressantworten. Bei der Entwicklung von Therapien mit Base Editoren ist jedoch die potenzielle Off-Target-Aktivität ein wichtiger Aspekt. Umfangreiche genomeweite Analysen mit Techniken wie CIRCLE-seq und Hochdurchsatzsequenzierungen haben gezeigt, dass zwar einige Off-Target-Effekte auftreten, diese meist in nicht-kodierenden Regionen liegen oder zu synonymeren Mutationen führen, die keine Veränderung des proteinischen Outputs bewirken. Dennoch ist eine gründliche Sicherheitsbewertung essentiell, insbesondere vor einer Translation in klinische Anwendungen. Zukünftige Entwicklungen könnten die Anwendung des Base Editings durch die Kombination mit verbesserten Editoren mit höherer Spezifität und neuen Vektorsystemen weiter verbessern.
So könnten unterschiedliche AAV-Serotypen oder andere Liefermethoden eingesetzt werden, um neben neuronalen Zellen auch weitere relevante Zielzellen wie Gliazellen oder Herzmuskelzellen anzusprechen, was beispielsweise für FRDA von großem Interesse ist, da diese Krankheit auch das Herz stark beeinträchtigt. Es ist anzumerken, dass gängige Tiermodelle, wie die Htt.Q111 oder YG8s Mäuse, nicht alle klinischen und neuropathologischen Merkmale der menschlichen Erkrankungen adäquat widerspiegeln. Die Forschung gewinnt daher zunehmend durch die Entwicklung neuer Modelle an Relevanz, die neben der molekularen auch die physiologische Ebene besser simulieren, um den translativen Wert der Base Editing-Strategien zu erhöhen. Die Hoffnung liegt darin, dass diese präzisen Editierungstechnologien das Fortschreiten von Huntington und Friedreich-Ataxie zumindest verzögern oder gar stoppen können.




![Inigo Quilez – Unlocking Creativity with Signed Distance Fields [video]](/images/7F813F89-C807-4A13-9D94-A0C1ABD962E6)
