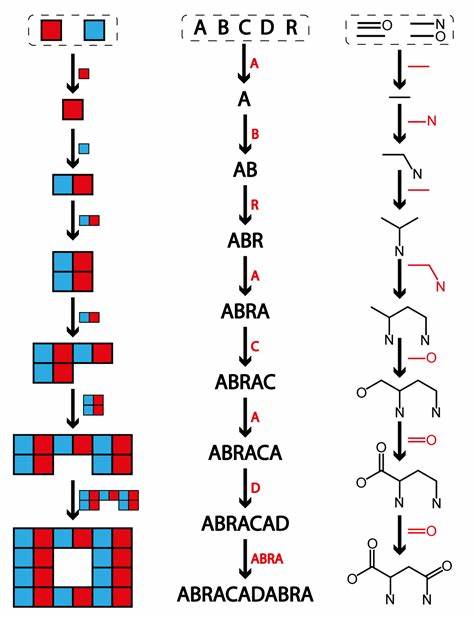
Die Assembly-Theorie gewinnt immer mehr an Bedeutung in der modernen Wissenschaft, insbesondere in der Chemie und Molekularbiologie. Zentraler Bestandteil dieser Theorie ist der Assembly Index, ein quantitativer Wert, der die Komplexität eines Moleküls anhand seiner Struktur beschreibt. Vereinfacht gesagt misst der Assembly Index die Mindestanzahl an Schritten, die notwendig sind, um die Molekülstruktur aufzubauen, ausgehend von einfacheren Bausteinen. Dieses Konzept revolutioniert das Verständnis der molekularen Synthese und bietet neue Perspektiven für Forschung und Anwendung. Was genau bedeutet der Assembly Index? Man kann sich das anhand eines alltäglichen Beispiels besser vorstellen: Nehmen wir ein Wort wie „ABRACADABRA“.
Obwohl dieses Wort neun Buchstaben umfasst, setzt es sich nur aus vier einzigartigen Buchstaben zusammen: A, B, C und R. Diese Buchstaben entsprechen den „Grundbausteinen“. Um das gesamte Wort zu konstruieren, beginnt man mit einfachen Kombinationen wie „AB“. Indem immer weitere Segmente kombiniert und wiederverwendet werden, gelangt man letztlich zum kompletten Wort. Entscheidend ist dabei die Wiederverwendung zuvor erstellter Strukturen, was die Anzahl der Schritte reduziert.
Das bedeutet, es geht darum, den effizientesten Weg zu finden, um das Zielobjekt, hier das Wort, aufzubauen. Auf molekularer Ebene bedeutet das, dass der Assembly Index die minimale Anzahl von Schritten repräsentiert, um die Struktur eines Moleküls zu formen - vor allem anhand seiner graphbasierten Darstellung. Diese Sichtweise unterscheidet sich bewusst von der tatsächlichen chemischen Synthese im Labor, da hier eher ein theoretisches Konstrukt betrachtet wird, das nicht zwangsläufig praktisch herstellbare Schrittfolgen darstellt. In diesem Rahmen werden Wasserstoffatome häufig ignoriert, da sie die strukturelle Komplexität kaum verändern und somit die Berechnung erheblich vereinfachen. Ein wesentliches Merkmal der Assembly-Theorie ist die Berücksichtigung von Bindungen als fundamentale Bauelemente anstelle einzelner Atome.
Das bedeutet, dass man bei der Konstruktion eines Moleküls davon ausgeht, dass Bindungen zwischen Atomen zusammengefügt werden. Diese Herangehensweise hilft, die molekulare Komplexität genauer und effizienter zu beschreiben. So entsteht ein klareres Bild von der Art und Weise, wie Moleküle theoretisch aufgebaut sind und wodurch sie sich unterscheiden. Das Berechnen des Assembly Index ist allerdings keine triviale Aufgabe. Denn die Komplexität von Molekülen und deren möglichen Verbindungswegen führt zu einem NP-Hard-Problem.
Das heißt, dass der Rechenaufwand exponentiell mit der Größe des Moleküls wächst. Für größere Moleküle wird es dadurch schnell schwierig, den Assembly Index direkt zu bestimmen. Deshalb greifen Wissenschaftler auf verschiedene algorithmische Methoden zurück, um Abschätzungen oder exakte Werte zu erhalten. Eine bewährte Methode zur Berechnung des Assembly Index ist der sogenannte Split-Branch-Algorithmus. Dieses Verfahren schafft es, eine obere Schranke für den Assembly Index zu bestimmen, indem es größere Teilstrukturen innerhalb eines Moleküls identifiziert und separat bewertet.
Dabei kann es allerdings vorkommen, dass Überschneidungen von Teilstrukturen zu einer Überzählung führen, was zu etwas höheren Werten führt als der tatsächlich minimal erforderliche Aufwand. Dennoch gilt dieser Algorithmus als effizient und praktikabel, insbesondere für komplexere Moleküle, bei denen eine exakte Berechnung zu rechenintensiv wäre. Die Entwicklung und Verbesserung solcher Algorithmen ist ein aktives Forschungsfeld, da man bestrebt ist, genauere und trotzdem rechenoptimierte Methoden zu finden. Neben dem Split-Branch-Algorithmus sind bereits Monte-Carlo-basierte Verfahren in Planung, um probabilistische Annäherungen an den tatsächlichen Assembly Index zu ermöglichen. Dadurch wird langfristig eine breitere Anwendung und schnellere Auswertung von umfangreichen Molekülbibliotheken ermöglicht.
Was macht den Assembly Index so wertvoll? Er stellt eine messbare Größe dar, mit der man Molekülkomplexität nicht nur bewerten, sondern auch miteinander vergleichen kann. Höhere Assembly Indices deuten auf Moleküle hin, die komplexer und damit potenziell schwerer herzustellen sind oder stärkere biologische Funktionen besitzen. In der Astrobiologie beispielsweise kann der Assembly Index verwendet werden, um mögliche Biosignaturen zu identifizieren – also Molekülstrukturen, die parallelen zu biologisch erzeugten Verbindungen zeigen und somit auf Leben als Quelle hinweisen könnten. Darüber hinaus bietet die Assembly-Theorie eine neue Perspektive auf die Erforschung präbiotischer Chemie und der Entstehung von Leben. Durch die Analyse der zugrundeliegenden molekularen Komplexität lässt sich nachvollziehen, welche Verbindungen unter welchen Bedingungen wahrscheinlich gebildet werden und wie sich komplexe organische Strukturen im Laufe der Zeit entwickelt haben könnten.
Die Ignorierung von Wasserstoffatomen bei der Berechnung des Assembly Index sollte dabei nicht als Einschränkung verstanden werden. Im Gegenteil, dieser oft praktizierte Schritt erlaubt es, sich auf die eigentliche Rückgratstruktur eines Moleküls zu konzentrieren, die seine Funktionalität und Form maßgeblich bestimmt. Dies beschleunigt nicht nur die Berechnungen, sondern führt auch zu aussagekräftigeren Ergebnissen in Bezug auf die Informationsdichte eines Moleküls. Die Anwendungsmöglichkeiten der Assembly Theory und des Assembly Index sind vielfältig und reichen weit über die Grundlagenforschung hinaus. In der Materialwissenschaft helfen diese Konzepte dabei, neue Verbindungen oder Polymerstrukturen zu entwerfen, deren Komplexität und Stabilität abschätzbar sind.
In der Pharmaforschung ermöglicht das Verständnis der molekularen Komplexität eine systematische Bewertung potenzieller Wirkstoffe und deren Synthesewege. Ein weiterer spannender Aspekt ist die Visualisierung von Molekülen als graphische Strukturen, die Schritt für Schritt aufgebaut werden. Dies führt nicht nur zu einem besseren Verständnis der molekularen Architektur, sondern erleichtert auch die Identifikation von wiederkehrenden Mustern oder Motiven innerhalb großer Moleküle – ein Ansatz, der sich auch in der Bioinformatik und beim Studium von Proteinen und Nukleinsäuren als hilfreich erweist. Die rechnergestützte Vorhersage von Assembly Indices wird heute zunehmend durch spezialisierte Datenbanken und Plattformen bereitgestellt, die vorab berechnete Werte zur Verfügung stellen. Dies entlastet Forscher von aufwendigen Berechnungen und fördert die schnelle Anwendung der Theorie auf neue Fragestellungen.