Die Frage, wie Moleküle zu Zeichen wurden, berührt die tiefgründigen Ursprünge des Lebens und die Grundlagen biologischer Information. Lange Zeit galt die Auffassung, dass Moleküle wie DNA und RNA schlicht Datenspeicher seien – reine Träger von genetischer Information, deren Rolle vor allem in der Speicherung und Weitergabe von Mustern lag. Doch dieser Ansatz übergeht eine entscheidende Dimension: die Bedeutung und Interpretation der Information, also die Semiotic, die Lehre von Zeichen und deren Deutung. Bereits Erwin Schrödinger stellte in seinem Werk „Was ist Leben?“ fest, dass es nicht ausreicht, lediglich die physikalisch-chemischen Eigenschaften von Molekülen zu betrachten, sondern dass Lebewesen durch eine Kombination aus energetisch fern vom thermodynamischen Gleichgewicht liegender Organisation und Informationsspeicherung charakterisiert sind. In der Folge wurde die genetische Information im Sinne des sogenannten Zentraldogmas der Molekularbiologie (DNA zu RNA zu Protein) verstanden, wobei Information häufig lediglich als Muster betrachtet wurde – inkompatibel mit einer systematischen Bedeutung oder einem „Über etwas Sein“ (Aboutness).
Das Problem bei diesem traditionellen Verständnis liegt in der Vernachlässigung von Interpretation. Ein Molekül an sich kann keine Bedeutung tragen; es ist lediglich eine physische Struktur. Für die Information als etwas Bedeutungsvolles ist eine interpretierende Instanz unerlässlich. Überträgt man dies auf biologische Systeme, so wird schnell klar, dass lediglich die dynamische Interaktion und Interpretation eines Moleküls in einem zellulären Kontext es zu einem Zeichen macht – es wird etwas über andere Moleküle „gesagt“. Dies ist eine fundamentale semiotische Einsicht: Es ist der interpretive Prozess, der den materiellen Eigenschaften erst Bedeutung gibt.
Um diesen komplexen Prozess zu verstehen, haben Wissenschaftler wie Terrence W. Deacon vorgeschlagen, mit einfachen molekularen Modellen zu arbeiten, die auf real physikalisch-chemischen Prozessen basieren, wie selbstorganisierende und rekursive Systeme. Ein Beispiel hierfür sind sogenannte „Autogene Viren“, hypothetische, sich selbst reproduzierende Virusähnliche Systeme, die auf den Prinzipien reziproker Katalyse und Selbstassemblierung beruhen. Diese Systeme bieten eine minimalistische Grundlage, um zu zeigen, wie molekulare Systeme Informationsverarbeitung und Interpretation bereits auf einfachster Ebene ausführen können. Reziproke Katalyse beschreibt hierbei ein System, in dem ein Katalysatorprodukt die Produktion eines anderen Katalysators fördert, wodurch sich eine kreisförmige Reaktionskette bildet.
Selbstassemblierung hingegen ist ein Prozess, bei dem Moleküle aufgrund ihrer Form- und Ladungseigenschaften spontan zu komplexen Strukturen wie Viruskapsiden zusammenfinden. Wenn diese beiden Prozesse gekoppelt werden, entsteht ein autogenes System, das sich selbst erhält, repariert und sogar reproduziert. Diese Dynamik liefert eine Vorlage, wie molekulare Systeme Bedeutung tragen können: Veränderungen im System, wie die Beschädigung einer Kapsel, wirken als signifikante Ereignisse (Zeichen), auf die das System mittels Interpretation reagiert, um sich wiederherzustellen. Ein weiteres zentrales Element wird in der Verschränkung von Arbeitszyklus und physikalischer Einschränkung gesehen. Arbeit kann nur ausgeführt werden, wenn es Einschränkungen gibt, die Energiefreisetzung steuern.
Gleichzeitig braucht es Arbeit, um diese Einschränkungen neu zu erzeugen und zu erhalten. Im molekularen Kontext bedeutet dies, dass ein autogenes System durch sein eigenes dynamisches Verhalten Constraints, also funktionale Schranken, reproduziert, die wiederum seine Existenz sichern. Die graduelle Steigerung der interpretativen Kompetenz eines solchen Systems führt zu einer semiotischen Entwicklung, die analog zu Peirces Theorie in Ikone, Index und Symbol gegliedert werden kann. Die einfachste Form, das ikonische Interpretieren, basiert auf Ähnlichkeit und Unterscheidungsvermögen: Das System differenziert zwischen intaktem und beschädigtem Zustand, interpretiert diese Differenz als Signale und reagiert darauf. Auf dieser Grundlage kann eine indexikale Interpretation entstehen – das Erkennen von Zusammenhängen mit Umweltzuständen beispielsweise durch Veränderungen auf der Oberfläche, die Bindungsmöglichkeiten für Substrate beeinflussen und so als Hinweise auf günstige Umweltbedingungen dienen.
Schließlich ermöglichen komplexere Systeme die symbolische Interpretation – das Abrufen und Übertragen von Referenzen, wie wir sie bei der DNA-gestützten Informationsspeicherung beobachten. Interessant ist hierbei die Einsicht, dass die selektive Aufnahme und Reproduktion solcher molekularer Systeme einen evolutiven Vorteil darstellt. Systeme, die Umweltinformationen effizient interpretieren und darauf reagieren, können ihre Reproduktion optimieren, wodurch sich semantisch sinnvolle Information durch natürliche Selektion herausbildet und verfestigt. Die Rolle der Nukleinsäuren wie RNA und DNA wird in diesem Kontext neu bewertet. Statt als initiale Informationsträger werden sie als semiotische Artefakte betrachtet, die physikalische und chemische Eigenschaften besitzen, welche es dem interpretierenden System ermöglichen, Informationen auf einem anderen Substrat zu kodieren und zu übertragen.
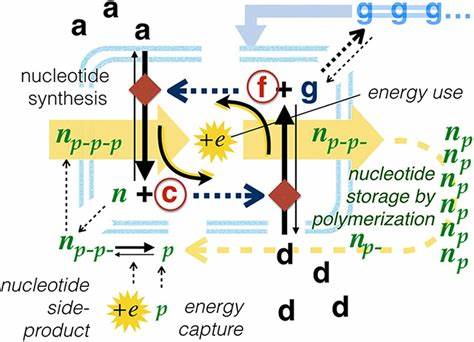
Dies erfolgt durch eine Art „Verlagerung“ oder „Offloading“ der dynamischen Constraints auf stabile, sich selbst replizierende Molekülstrukturen, wodurch die Komplexität der Informationsverarbeitung maschinell erweitert wird. Die Tatsache, dass Nukleotide duale Funktionen besitzen – sowohl im Energiestoffwechsel als auch als Bausteine für Informationspolymere – unterstreicht ihre evolutionäre Relevanz für die Entstehung biologischer Semantik. Parallel zur Entwicklung dieser molekularen Informationssysteme wächst die semiotische Komplexität in Organismen, die Schichtungen von Interpretationsprozessen erzeugen, die aufeinander aufbauen: von molekularen zu zellulären, dann zu gewebespezifischen und schließlich zu organismischen oder sozialen Zeichenebenen. Dieses Prinzip lässt sich als semiotisches Scaffolding beschreiben, das die Evolution ermöglicht, sich von einfachen interpretativen Mechanismen zu hochkomplexen semantischen Systemen zu entwickeln. Die moderne Biosemiotik verknüpft dabei Erkenntnisse der Molekularbiologie mit Theorien der Semiotik und Systemtheorie, um die Entstehung von Sinnstrukturen im Lebendigen besser zu verstehen.
Zentral ist die Erkenntnis, dass biologische Information niemals losgelöst von den interpretierenden und funktionalen Prozessen betrachtet werden kann. Ohne ein interpretierendes System, das molekulare Muster in funktionale Bedeutungen übersetzt, bleibt Information Leerform ohne Bezug. In der Konsequenz wird das Verständnis der Information in biologischen Systemen grundlegend erweitert: Information wird zu einem Produkt des dynamischen Zusammenspiels von Constraint, Arbeit und Interpretation. Moleküle werden zu Zeichen, wenn sie im Kontext eines selbstorganisierenden Systems auf eine Art verwendet werden, die Funktion und Bedeutung erzeugt. Diese Perspektive führt zu einem Umdenken in der Biologie: Die klassischen Modelle, die DNA und RNA als Beginn der biologischen Information ansehen, erscheinen unvollständig.