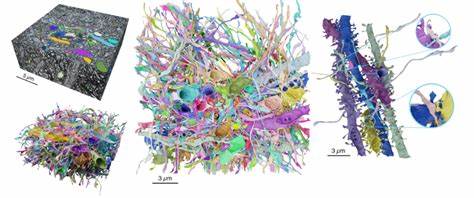
Die Struktur und Funktion des Gehirns basieren maßgeblich auf der komplexen Vernetzung seiner Milliarden von Neuronen, die über feinste synaptische Verbindungen miteinander kommunizieren. Diese neuronale Verschaltung oder „Connectom“ ist die Grundlage für sämtliche kognitiven Prozesse, von Wahrnehmung über Lernen bis hin zu Gedächtnis. Ein detailliertes Verständnis dieses Verbindungsnetzwerks im Gehirn von Säugetieren war lange Zeit eine Herausforderung, vor allem wegen der begrenzten räumlichen Auflösung herkömmlicher bildgebender Verfahren und der Schwierigkeit, molekulare Informationen mit strukturellen Daten zu verbinden. Hier setzt die lichtmikroskopische konnektomische Rekonstruktion an, ein innovatives Verfahren, das mithilfe von Lichtmikroskopie und Hydrogel-gestützter Probenausdehnung eine synapsenauflösende Abbildung des Gehirngewebes ermöglicht. Lichtmikroskopie als Schlüsselwerkzeug und ihre Limitationen Die traditionelle Lichtmikroskopie bietet den Vorteil, dass sie spezifische Moleküle mittels Fluoreszenzmarkierung sichtbar machen kann.
Allerdings liegt die Limitierung in ihrer räumlichen Auflösung, die durch die Beugungsgrenze des Lichts bei etwa 200 bis 300 Nanometern lateral und erheblich größer im axialen Bereich liegt. Angesichts der Dichte des neuronalen Gewebes und der winzigen Dimensionen von Synapsen, welche oft im Bereich von weniger als 100 Nanometern liegen, war es bislang unmöglich, komplette synapsenpräzise Verschaltungen dicht betonter Nervenzellverbände ausschließlich mit Lichtmikroskopie zu rekonstruieren. Demgegenüber liefert die Elektronenmikroskopie (EM) eine Auflösung im Nanometerbereich, mit der synaptische Strukturen und Verknüpfungen detailliert abgebildet werden können. Dennoch bringt EM mehrere Nachteile mit sich: die Probenvorbereitung ist aufwendig, die molekulare Charakterisierung einzelner Synapsen nur eingeschränkt möglich, und die Datenerhebung sowie -auswertung sind extrem ressourcenintensiv. Die Kombination von EM mit korrelativer Lichtmikroskopie ist zwar möglich, stellt aber einen hohen technischen Aufwand dar und ist meist auf kleine Volumina beschränkt.
Hydrogel-gestützte Probenexpansion als Innovation Eine wegweisende Methode zur Umgehung der Begrenzungen der Lichtmikroskopie ist die sogenannte Expansion Microscopy (ExM). Dabei wird das biologische Gewebe in ein Polyacrylamid-Hydrogel eingebettet und anschließend chemisch behandelt, sodass das Hydrogel aufquillt und das Gewebe linear auf ein Vielfaches seiner originalen Größe erweitert wird. Durch die Expansion vergrößern sich die Abstände zwischen Strukturen, sodass sie mit konventionellen Lichtmikroskopen in einer für Synapsen ausreichenden Detailgenauigkeit dargestellt werden können. Das von Forschenden neu entwickelte LICONN (Light-microscopy-based Connectomics) baut auf dieser Idee auf und verwendet eine mehrstufige, iterative Hydrogel-Expansion. Zunächst erfolgt eine vierfache Expansion gefolgt von der Polymerisation eines nicht ausdehnbaren Stabilisators und einer zweiten Expansionsstufe, welche das Volumen um den Faktor circa 16 vergrößert.
Dieses Verfahren beinhaltet eine innovative chemische Fixierung und Verankerung der Proteine an das Hydrogel mittels multifunktionaler Epoxide, was die Zellstrukturen hochpräzise bewahrt und ein Höchstmaß an struktureller Integrität garantiert. Hohe Auflösung bei großer Probengröße Mit LICONN wird die Lichtmikroskopie auf eine effektive Auflösung von rund 20 Nanometern lateral und 50 Nanometern axial gebracht, wodurch synaptische Strukturen detailliert sichtbar werden. Zugleich können ausgedehnte Gewebevolumina von etwa einem Kubikmillimeter erfasst werden, was repräsentativ für eine substanzielle Hirnregion ist. Detaillierte Analyse neuronaler Netzwerke Die ausgefeilte Probenvorbereitung erlaubt es, selbst die dünnsten Axone und Dendriten mit ihren spitzen Verzweigungen nachzuverfolgen. Die manuelle und automatische Nachverfolgung dieser Strukturen auf Basis der LICONN-Daten liefert eine Verbindungstreue, mit Fehlerquoten bei der Routenbestimmung, die mit denen in der Elektronenmikroskopie vergleichbar sind.
Erklärtes Ziel ist die vollständige, dichte Rekonstruktion neuronaler Schaltkreise inklusive der feinsten Prozesse, was durch den Einsatz leistungsfähiger, tiefenlernender Algorithmen (bspw. Flood-Filling Networks) realisiert wird. Integration molekularer Marker Ein besonders wichtiger Fortschritt von LICONN ist die zeitgleiche molekulare Charakterisierung der struktu- rellen Daten. Post-expansionsimmunfärbungen erlauben spezifische Markierung von Synapsenproteinen sowohl auf der präsynaptischen als auch auf der postsynaptischen Seite. So können etwa Bassoon als präsynaptischer Marker und SHANK2 beziehungsweise PSD95 als postsynaptische Marker mit hoher Genauigkeit detektiert werden.
Diese molekularen Marker ermöglichen die Unterscheidung von exzitatorischen und inhibitorischen Synapsen sogar auf der Ebene einzelner Kontaktstellen. Nebenchemische Synapsen können auch elektrische Synapsen (Gap Junctions) durch den spezifischen Nachweis von Connexin-43 analysiert werden. Somit kann LICONN das Gehirn nicht nur als Netz von Verschaltungen abbilden, sondern deren funktionelle (exzitatorisch, inhibitorisch, elektrisch) und molekulare Eigenschaften mit erfassen. Automatisierte Erkennung synaptischer Verbindungen Die Kombination aus hochauflösenden strukturellen Aufnahmen und molekularen Signalen ermöglicht nicht nur die visuelle Validierung, sondern auch die Entwicklung automatisierter Algorithmen zur Synapsenerkennung. Ein ausgeklügelter Workflow ordnet immunmarkierte prä- und postsynaptische Strukturen zu vollständigen Synapsen zusammen und unterscheidet auch komplexe Verschaltungsarten, beispielsweise eine präsynaptische Struktur, die mehrere postsynaptische Partner hat.
Darüber hinaus erlauben tiefenlernende neuronale Netzwerke die Vorhersage synaptischer Molekülstandorte allein anhand der strukturellen Bilddaten. Damit ist die Analyse molekularer Synapseneigenschaften auch möglich, ohne für alle Marker direkte Immunfärbungen durchführen zu müssen – ein enormer Vorteil für großvolumige Datensätze. Vielfältige Anwendungen der Technologie LICONN eröffnet eine Reihe neuer Einsatzmöglichkeiten in der Hirnforschung. So erlaubt sie die Quantifizierung der Dichte exzitatorischer und inhibitorischer Eingänge auf Dendriten sowie die Identifikation spezifischer Zelltypen durch Marker wie Somatostatin oder KV3.1b.
Die Methode ermöglicht auch die Darstellung und Messung subzellulärer Strukturen wie primärer Zilien, die wichtige Signalzentren sind, und die Analyse deren Veränderungen bei genetischen Modellen von neurologischen Erkrankungen. Zudem kann die myelinierte Axonstruktur sowie die Verteilung von Gap Junctions (elektrische Synapsen) visualisiert werden, was bisher nur sehr schwer möglich war. Skalierbarkeit und praktische Anwendung Ein entscheidender Vorteil von LICONN ist die Nutzung allgemein verfügbarer, diffraction-limitierter Lichtmikroskope, wie Spinning-Disk-Konfokalgeräte, die weit verbreitet und vergleichsweise kostengünstig sind. Durch die mechanische Robustheit der expandierten Hydrogels können große Gewebevolumina in dünnen Scheiben iterativ erfasst und überlappend zusammengefügt werden, was eine Verlängerung der Analysezone in axialer Richtung ermöglicht. Die Verfahrensschritte für Hydrogel-Polymerisation, Fixierung, Labeling und Expansion sind gut dokumentiert und anwendbar, was eine breite Adoption der Methode in Standardlabors erlaubt.
Der Einsatz offener Deep-Learning-Softwarepakete zur Segmentierung und Analyse senkt weitere Hürden und ermöglicht auch Forschenden ohne tiefgehende Computerlinguistik-Kenntnisse die Nutzung der Technik. Perspektiven LICONN stellt einen vielversprechenden Durchbruch für die Neurowissenschaften dar, indem es erstmals eine umfassende und molekular informierte Niveau-synaptische Rekonstruktion großer Hirnareale mithilfe von Lichtmikroskopie ermöglicht. Damit schließt es die Lücke zwischen der hochaufgelösten, aber molekular limitierten EM-Technologie und der molekularreicheren, aber hinsichtlich der Genauigkeit limitierten herkömmlichen Lichtmikroskopie. Diese Methode bietet eine neue Grundlage, um zelluläre Netzwerke in Gesundheit und Krankheit besser zu verstehen, z.B.
die Defekte in der Synapsenvernetzung bei Entwicklungsstörungen oder neurodegenerativen Erkrankungen. Weiterhin kann die Kombination von LICONN mit in situ Transkriptionsanalysen zukünftig auch umfassende multimodale Karten von Zelltypen, Verbindungen und molekularen Profilen schaffen. Zusammenfassend eröffnet die lichtmikroskopische, hydrogelbasierte konnektomische Rekonstruktion einen neuen Weg zur Visualisierung und Analyse der komplexen neuronalen Architekturen im Gehirn von Säugetieren. Durch die Kombination aus hoher räumlicher Auflösung, molekularer Spezifität, Skalierbarkeit und Zugänglichkeit dürfte diese Technologie die Grundlagenforschung stark bereichern und neue Einsichten in die Funktionsweise des Gehirns ermöglichen.