Die Erforschung des menschlichen Genoms hat in den letzten Jahrzehnten enorme Fortschritte gemacht, doch erst die letzten Jahre haben die Komplexität der Genregulation durch die räumliche Organisation des Genoms in den Vordergrund gerückt. Insbesondere bei Krebs eröffnet das Verständnis der dreidimensionalen (3D) Architektur des Genoms neue Perspektiven auf die Entstehung, Progression und Behandlung von Tumoren. Die dreidimensionale Anordnung der DNA im Zellkern beeinflusst maßgeblich, wie Gene aktiviert oder stillgelegt werden – ein Prozess, der in Krebszellen häufig gestört ist und zur Deregulierung von Onkogenen und Tumorsuppressorgenen führt. Der Zellkern eines Menschen beinhaltet etwa zwei Meter DNA, die auf kleinstem Raum gefaltet und organisiert ist. Diese Faltung weist eine hierarchische Struktur auf, die von großen Chromosomenkompartimenten bis zu kleineren Bereichen, wie Topologically Associating Domains (TADs), reicht.
Innerhalb dieser Domänen finden sich dann spezifische Wechselwirkungen zwischen Distalen Regulatoren, sogenannten Enhancern, und den Promotoren der Zielgene. Dieser Mechanismus ist essentiell, damit Zellen unterschiedliche Gene je nach Typ und Funktion gezielt ein- oder ausschalten können. Krebszellen zeigen oftmals umfangreiche genomische Veränderungen, die nicht nur Mutationen in Genen selbst beinhalten, sondern auch strukturelle Variationen wie Translokationen, Inversionen oder Amplifikationen, die das 3D-Genom grundlegend verändern können. Solche Veränderung können sogenannte Enhancer-Rewirings hervorrufen, wodurch neue oder aberrante Genregulationsnetzwerke entstehen. So kann ein Enhancer, der normalerweise ein anderes Gen reguliert, durch diese strukturellen Veränderungen plötzlich ein Onkogen aktivieren, was die Entstehung von Krebs fördert.
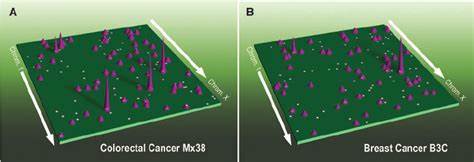
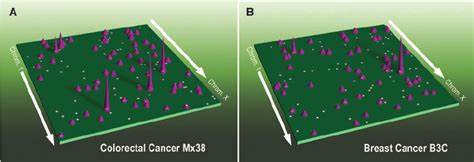
Ein bahnbrechendes Forschungsprojekt hat die 3D-Genomlandschaft von 69 Tumorproben aus 15 unterschiedlichen primären menschlichen Krebsarten analysiert. Hierbei wurde die HiChIP-Technologie eingesetzt, eine spezialisierte Chromatin-Konformationsmethode, die den Nachweis von DNA-Interaktionen mit gleichzeitiger Messung von H3K27ac (einem Markers für aktive Enhancer) ermöglicht. Die Kombination dieser Daten mit Ganzgenomsequenzierung und Einzelzellchromatinzugänglichkeit ermöglichte ein bisher unerreichtes Verständnis der Chromosomenarchitektur in Tumoren und deren mikrobiologischem Umfeld. Die Forschung brachte drei grundlegende Typen der Enhancer-Nutzung bei über 100 Onkogenen zum Vorschein: statische Nutzung, selektive Aktivierung und dynamisches Rewiring. Statische Enhancer sind krebsübergreifend aktiv und unterstützen die Grundregulation von Onkogenen.
Selektive Aktivierung zeigt sich zumeist in einem bestimmten Krebs-Typ, während dynamisches Rewiring eine sehr variable und oft patientenspezifische Veränderung in der Enhancer-Promotor-Kommunikation beschreibt. Besonders interessant war die Entdeckung, dass extrachromosomale DNA (ecDNA), kleine ringförmige DNA-Stücke außerhalb der Chromosomen, zu besonders intensiven Veränderungen der Enhancerlandschaft führt und damit Onkogene drastisch hochreguliert. Die dreidimensionale Organisation des Genoms erklärt auch, warum manche somatische Mutationen in nicht-kodierenden Regionen des Genoms, also in sogenannten regulatorischen Elementen, eine starke Auswirkung auf die Tumorentstehung haben können. Mutationen in Enhancer-Regionen, die durch HiChIP sichtbar gemacht wurden, zeigen häufig eine veränderte Bindung von Transkriptionsfaktoren und beeinflussen die Zielgenexpression maßgeblich. Durch integrative Analysen konnten diese Mutationen oft direkt mit veränderten Enhancer-Promoter-Interaktionen verknüpft werden.
Ebenso wurden die Auswirkungen struktureller Variationen auf die 3D-Genomorganisation detailliert beleuchtet. Während einfache chromosomale Rearrangements wie Deletionen, Duplikationen, Inversionen oder Translokationen bereits erhebliche Änderungen der Genregulation hervorrufen können, wirken komplexe Veränderungen, wie chromothripsis oder breakage-bridge fusion Zyklen, oft noch umfassender. Insbesondere ecDNA zeigt in der Studie ein hohes Maß an neuer Vernetzung zwischen Enhancern und Promotoren, was zu einer massiven Deregulierung von Onkogenen führt. Die Erkenntnisse aus der 3D-Genomanalyse werfen auch ein Licht auf die Bedeutung des Tumormikromilieus (TME), also das unmittelbare Umfeld von Tumorzellen, bestehend aus Immun-, Stützzellen und weiteren Zelltypen. Durch die kombinatorische Nutzung einzelzelliger Chromatinzugänglichkeit (scATAC-seq) und HiChIP konnten zelltypspezifische Enhancer-Promotor-Interaktionen identifiziert werden.
So zeigen beispielsweise myeloide Immunzellen spezifische Enhancer-Promotor-Schleifen für Gene wie CD274 (PD-L1), die eine Rolle bei der Immunflucht von Tumorzellen spielen. Diese zelltypspezifischen Interaktionen stellen wichtige potenzielle therapeutische Ansatzpunkte dar, da sie die komplexe Regulation der Immunantwort im Tumor widerspiegeln. Die umfassende Analyse der 3D-Genomstruktur bietet nicht nur neue Einsichten in die molekularen Grundlagen der Genregulation in Krebs, sondern eröffnet auch weitreichende Möglichkeiten für diagnostische und therapeutische Innovationen. Beispielsweise können Muster im Enhancer-Rewiring oder in der ecDNA-Bildung als Biomarker für den Krankheitsverlauf und Therapieansprechen dienen. Die Identifizierung funktionaler nicht-kodierender Mutationen kann die Entwicklung präziser medizinischer Interventionen unterstützen.
Darüber hinaus zeigen die Ergebnisse, dass die klassische Analyse von Genkopienzahlen (Copy Number Variation) zwar einen wichtigen Beitrag zur Erklärung der Genexpression in Tumoren leistet, die Aktivität von Enhancern jedoch häufig eine noch größere Rolle spielt. Für viele Onkogene ist die Expression stärker durch veränderte Enhancernetzwerke geprägt als durch simple Genamplikationen. Dies verdeutlicht die Komplexität der epigenetischen und strukturellen Regulation in Krebs und unterstreicht die Notwendigkeit integrierter multi-omics Ansätze. Die gewonnenen Daten wurden in Einklang mit früheren Studien gebracht, bestätigen und erweitern bestehendes Wissen über die stabilen strukturellen Kompartimente und dynamischen Enhancer-Netzwerke des Genoms. Die vielfach beobachtete Erhaltung der A/B-Kompartimente und TADs in Tumoren legt nahe, dass diese Strukturen als Grundgerüst fungieren, innerhalb dessen die fein abgestimmten Enhancer-Promotor-Wechselwirkungen für differenzierte Expressionsmuster sorgen.
Die Anwendung von Technologien wie HiChIP in Kombination mit tiefgreifender Sequenzierung und Einzelzellanalysen repräsentiert einen bedeutenden Fortschritt für die Krebsforschung. Sie ermöglicht zum ersten Mal systematische Einblicke in die dreidimensionale Organisation des Krebsgenoms direkt an Primärtumoren, anstatt nur auf Zelllinien oder tierische Modelle angewiesen zu sein. Dies führt zu einer realitätsnäheren Abbildung der molekularen Vorgänge, die die Tumorbiologie steuern. Aus regulatorischer Sicht sind insbesondere die dynamischen und selektiven Enhancer-Netzwerke spannend, da sie auch die Grundlage für das Phänomen der Tumorheterogenität bilden. Patientenspezifische Unterschiede in der 3D-Genomorganisation können erklären, warum unterschiedliche Patienten auf dieselben Therapien unterschiedlich ansprechen und warum Krebszellen oft schnell Resistenzen entwickeln.
Schließlich liefert die Untersuchung extrachromosomaler DNA-Moleküle neue Einblicke in die evolutionäre Dynamik von Tumoren. Die ecDNAs entziehen sich meist den klassischen Mechanismen der Chromosomenorganisation und -segregation und können dadurch Onkogene überproportional amplifizieren und spontan neue regulatorische Interaktionen erzeugen. Diese Erkenntnisse eröffnen neue Forschungsfelder im Bereich der epigenetischen Modulation und der zielgerichteten Therapie von ecDNA-assoziierten Krebserkrankungen. Zusammenfassend lässt sich sagen, dass die detaillierte Analyse der dreidimensionalen Genomlandschaft in primären menschlichen Krebsarten unser Verständnis der Tumorentstehung und -progression wesentlich erweitert. Die Bedeutung der 3D-Genomorganisation für die Genregulation im Krebs macht sie zu einem vielversprechenden Ansatzpunkt für zukünftige diagnostische Methoden und Therapien.
Die Kombination aus genetischen, epigenetischen und strukturellen Daten auf Einzelzellebene ermöglicht es, Krebs nicht nur als genetische, sondern als dreidimensionale epigenetische Erkrankung zu begreifen. Die daraus resultierenden Erkenntnisse werden maßgeblich dazu beitragen, die Präzision und Wirksamkeit von Krebstherapien im Zeitalter der personalisierten Medizin zu verbessern.