Die Erforschung von krankhaften Geweben stellt die Wissenschaft vor große Herausforderungen. Während traditionelle Methoden oftmals nur einzelne Zelltypen isoliert betrachten, ermöglichen innovative Ansätze zunehmend die Analyse der räumlichen Verteilung und der Interaktionen zwischen Zellen direkt im Gewebe. Ein bahnbrechendes Beispiel dafür ist die neue computergestützte Methode namens MESA (Multiomics and Ecological Spatial Analysis), die von einem interdisziplinären Forscherteam aus führenden Institutionen wie MIT, Stanford University und dem Broad Institute entwickelt wurde. MESA revolutioniert die Erforschung von Erkrankungen, indem es Gewebe als komplexe Ökosysteme betrachtet und auf diese Weise versteckte Muster in der Zellorganisation und -vielfalt sichtbar macht. Die Methode kombiniert ökologische Konzepte mit räumlicher Omics-Technologie und bietet dadurch einen völlig neuen Blick auf die Dynamik und Progression von Krankheiten.
Lange Zeit war die Herausforderung bei der Analyse von krankem Gewebe, dass Wissenschaftler meist isolierte Zelltypen oder molekulare Marker untersuchten, ohne die räumliche Einbettung und die Wechselwirkungen der Zellen zu berücksichtigen. Doch gerade diese „Nachbarschaften“ oder räumlichen Muster innerhalb des Gewebes spielen eine entscheidende Rolle bei der Krankheitsentwicklung, dem Fortschreiten und der Reaktion auf Therapien. Die MESA-Methode nutzt deshalb die Erkenntnisse der Ökologie, die bisher vor allem in der Umweltforschung zur Beschreibung von Biodiversität und Artenverteilung angewandt wurden, um zelluläre Biodiversität und räumliche Interaktionen im Gewebe zu kartieren und zu quantifizieren. MESA basiert auf der Analyse von sogenannten spatialen Omics-Daten, das heißt, es verarbeitet molekulare Daten, wie die Genexpressionsprofile oder Proteinlevel verschiedener Zelltypen, die gleichzeitig mit ihrer genauen Position im Gewebe erfasst werden. Moderne Techniken wie spatial transcriptomics oder proteomics ermöglichen es, diese hochdetaillierten Informationen zu gewinnen, die traditionelle Sequenzierungsmethoden weit übertreffen.
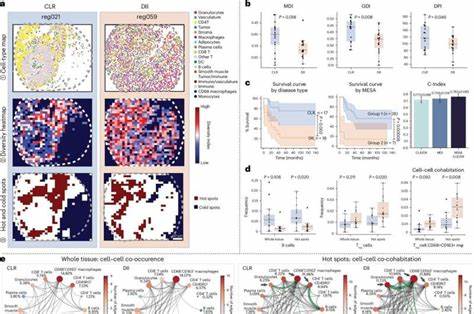
Dennoch stellte die immense Datenmenge mit komplexen räumlichen Mustern eine Herausforderung für die Datenanalyse dar – hier setzt MESA an. Durch die Integration mehrerer Omics-Datenebenen in Kombination mit robusten ökologischen Modellen wird ein vielschichtiges Bild der Gewebearchitektur geschaffen. Durch die Anwendung von Prinzipien aus der Ökologie, etwa dem Studium von Habitatfragmentierung oder Arteninteraktionen, versucht MESA, Ähnlichkeiten zwischen Zellpopulationen und biologischen Arten zu erkennen und deren räumliche Verteilung im kranken Gewebe zu verstehen. So können etwa „biologische Hotspots“ identifiziert werden – Bereiche, in denen spezifische Zelltypen besonders häufig zusammen auftreten und möglicherweise entscheidenden Einfluss auf die Krankheitsentwicklung haben. Diese Erkenntnisse eröffnen völlig neue Perspektiven, etwa beim Verständnis der Tumormikroumgebung im Krebs oder der chronischen Entzündungsprozesse bei Autoimmunerkrankungen.
Ein anschauliches Beispiel für die Leistungsfähigkeit von MESA liefert die Analyse von Leberkrebsproben. Das Verfahren deckte Regionen auf, in denen Tumorzellen regelmäßig zusammen mit Makrophagen vorkommen. Diese Zellzusammensetzung könnte darauf hinweisen, dass solche Zonen eine besondere Rolle bei der Progression oder der Resistenz gegen Therapien spielen. Solche Ergebnisse könnten künftig dazu beitragen, gezieltere Behandlungsstrategien zu entwickeln oder präzise diagnostische Marker zu identifizieren. Das Verständnis der räumlichen Interaktionen von Immunzellen und Tumorzellen wächst damit zu einem wichtigen Baustein für personalisierte Medizin.
Neben der Entdeckung neuer räumlicher Muster bietet MESA eine weitere wesentliche Innovation: die Möglichkeit der computationalen Verfeinerung von Gewebedaten. Anstatt kostspielige und komplexe neue Experimente durchzuführen, kann die Methode bereits existierende Datensätze ergänzen, indem sie zusätzliche molekulare Informationen mittels Ki-Transfer aus öffentlich verfügbaren Einzelzell-Daten hinzufügt. Dies erweitert die Aussagekraft vorhandener Gewebeanalysen erheblich. So können Forscher nicht nur einzelne Zelltypen besser charakterisieren, sondern auch funktionale Zustände und molekulare Prozesse in räumlichem Kontext verstehen. Das Potenzial von MESA erstreckt sich weit über die Krebsforschung hinaus.
In zahlreichen weiteren Krankheitsbereichen – von Autoimmunerkrankungen bis hin zu neurologischen Leiden – könnten durch die Analyse der räumlichen Zellökologie neue Krankheitsmechanismen entdeckt werden. Durch die Verknüpfung verschiedener Omics-Ebenen, etwa von Transkriptom- und Proteom-Daten, entsteht ein umfassendes Bild, das die Komplexität des Gewebes als lebendes System widerspiegelt. Dies fördert nicht nur das grundlegende Verständnis von Krankheitsprozessen, sondern unterstützt auch die Entwicklung von Diagnostika und Therapeutika, die konkreter auf die räumliche Organisation im Gewebe abgestimmt sind. Mit der Veröffentlichung der MESA-Software als Open-Source-Python-Paket steht einer breiten Forschungsgemeinschaft ein leistungsfähiges Werkzeug zur Verfügung, das in akademischen Einrichtungen ebenso wie in der translationalen Forschung eingesetzt werden kann. Obwohl die räumliche Omics-Technologie derzeit noch nicht für den routinemäßigen Klinikalltag geeignet ist, gewinnt sie bereits in der pharmazeutischen Industrie an Bedeutung, insbesondere bei der Begleitung von Wirkstoffstudien, bei denen die Reaktionen des Gewebes auf Therapien entscheidend analysiert werden müssen.
Alex K. Shalek, einer der leitenden Wissenschaftler hinter MESA, hebt hervor, wie der interdisziplinäre Ansatz die Grenzen der Forschung verschiebt: Indem Methoden aus der Ökologie und modernster Molekularbiologie verbunden werden, könne man Gewebe nicht länger nur als Ansammlung einzelner Zellen sehen, sondern als dynamisches, räumlich organisiertes System mit eigenen Regelmechanismen. Die neue Perspektive ermögliche es, frühe Krankheitssignale zu erkennen und die Reaktion auf Therapien besser vorherzusagen. Bokai Zhu, Postdoktorand am MIT und Mitautor der Studie, beschreibt die Faszination, die von der Übertragung ökologischer Konzepte auf die Biomedizin ausgeht. Die Idee, Zellen wie Tierarten zu analysieren, eröffnet zahlreiche Möglichkeiten, um die oft verborgene Komplexität in kranken Geweben sichtbar zu machen.
Es sei ein neues Kapitel, das gerade erst begonnen habe – mit der Aussicht, Krankheit nicht nur besser zu verstehen, sondern auch effektiver zu bekämpfen. Insgesamt steht MESA für eine bemerkenswerte Innovation in der biomedizinischen Forschung. Es ist ein Zeichen dafür, wie interdisziplinäre Ansätze und modernste Technologien neue Wege eröffnen, komplexe medizinische Herausforderungen anzugehen. Die verborgene Ökologie der kranken Gewebe wird durch diese Methode nicht länger ein unbeachtetes Rätsel sein, sondern wird systematisch entschlüsselt – mit dem Ziel, die Behandlung vieler schwerwiegender Erkrankungen nachhaltig zu verbessern und die Präzisionsmedizin voranzutreiben.





![Radiance Fields and the Future of Generative Media [video]](/images/6D7BC1BB-452F-459E-9CDC-1D99FF45C25C)
