Die Untersuchung der dreidimensionalen Genomlandschaft bei primären menschlichen Krebserkrankungen hat in den letzten Jahren wesentlich an Bedeutung gewonnen. Anders als das einfache lineare DNA-Modell zeigt das Genom innerhalb einer Zelle eine komplexe räumliche Faltung und Organisation, die verschiedene Funktionsbereiche und Regulationsmechanismen ermöglicht. Diese dreidimensionale Architektur ist unerlässlich für die Kontrolle der Genaktivität, insbesondere durch die Interaktion von Promotoren mit entfernten Enhancern, welche die Genexpression in Gewebe- und Zelltypspezifischer Weise modulieren. Krebszellen zeigen oftmals tiefgreifende Veränderungen in dieser 3D-Anordnung, welche sowohl die Tumorentstehung als auch die Progression beeinflussen können. Das Verständnis der 3D-Genomstruktur eröffnet somit neue Perspektiven zum Verständnis der molekularen Mechanismen bei Krebs und zur Entwicklung gezielter Therapieansätze.
Innerhalb des Zellkerns ist die fast zwei Meter lange DNA hochkompakt in einem etwa zehn Mikrometer großen Raum untergebracht. Diese Kompaktheit wird durch eine komplexe und hierarchische Organisation ermöglicht. Genomabschnitte sind grundsätzlich in sogenannte A- und B-Kompartimente unterteilt, die aktive beziehungsweise inaktive Regionen des Genoms charakterisieren. Die A-Kompartimente enthalten überwiegend offene Chromatinstrukturen mit aktivem Genexpressionspotenzial, während B-Kompartimente für kompaktere, inaktive Chromatinbereiche stehen. Auf einer kleineren Skala bilden sogenannte Topologisch assoziierte Domänen (TADs) Megabase große Bereiche, in denen DNA-Sequenzen bevorzugt interagieren und die die räumliche Trennung unterschiedlicher Genregulationskomplexe unterstützen.
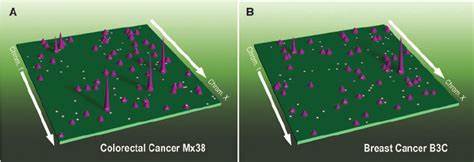
Auf der feinsten Ebene verbinden Enhancer-Promotor-Schleifen weit entfernt liegende regulatorische Elemente direkt mit ihren Zielgenen, was eine besonders präzise Steuerung der Genaktivität ermöglicht. Bei primären menschlichen Tumorerkrankungen lassen sich Veränderungen auf allen Ebenen der dreidimensionalen Genomorganisation nachweisen. Studien mit mehr als 60 Tumorproben aus 15 verschiedenen Krebspatientengruppen zeigten die Vielfalt der 3D-Genomlandschaften. Besonders auffällig sind Unterschiede in der Nutzung von Enhancern, die als Schlüsselregulatoren der Genexpression fungieren und bei Krebserkrankungen deutlich umgeschaltet werden können. Forscher konnten drei archetypische Muster der Enhancer-Nutzung über mehr als 100 Onkogene hinweg identifizieren: eine statische Nutzung, selektive Aktivierung bestimmter Enhancer in speziellen Krebsarten und eine dynamische Umstrukturierung der Enhancer-Verbindungen.
Letztere verdeutlicht, wie Genregulation in Tumoren durch die räumliche Neukonfiguration genetischer Elemente flexibel an Umweltbedingungen und molekulare Veränderungen angepasst wird. Ein zentraler Aspekt der 3D-Genomforschung im Kontext von Krebs ist die Analyse von strukturellen Varianten (SVs). Diese umfassen verschiedene Doppelstrangbrüche und Umlagerungen im Genom, darunter Duplikationen, Deletionen, Inversionen und Translokationen, die das Genom in seiner dreidimensionalen Struktur beeinflussen. Komplexe genomische Instabilitäten, wie etwa Chromothripsis und sogenannte extrachromosomale DNA (ecDNA), ein Kreisförmiges DNA-Element, das Onkogene massiv amplifizieren kann, führen zu einer verstärkten Umgestaltung der Genregulation. EcDNAs sind besonders bemerkenswert, da sie Onkogene außerhalb der chromosomalen Architektur platzieren, was zu einer verstärkten Genexpression und aggressiveren Tumorcharakteristika beiträgt.
Die Kombination aus Deep-Genomsequenzierung und Chromosomenkonformationsanalyse half dabei, solche strukturellen Veränderungen genau zu kartieren und ihre Auswirkungen auf die Enhancer-Regulation zu verstehen. Die Bedeutung der 3D-Genomorganisation zeigt sich auch darin, dass diese Architektur die Effekte von Punktmutationen in nichtkodierenden Regionen mit einbezieht. Solche Mutationen können etwa durch die Schaffung neuer Transkriptionsfaktor-Bindungsstellen zu einer Aktivierung von bislang inaktiven Enhancern führen. Die Folge sind veränderte Geneinstellungen bei Krebserkrankungen, was eine wichtige Dimension für die Erforschung nichtkodierender genomischer Mutationen darstellt. Durch die Zuordnung solcher Mutationen zu ihren Zielgenen über die dreidimensionale Genomstruktur können neue, bislang unbekannte treibende Mechanismen der Tumorentstehung aufgedeckt werden.
Neben den tumorzelleigenen Veränderungen beleuchtet die Forschung auch die Anlage des tumorumgebenden Mikromilieus (TME). Dieses Milieu besteht aus vielfältigen Zelltypen wie Immunzellen, Fibroblasten und Endothelzellen, die interagierend zur Tumorprogression beitragen oder diese hemmen können. Die Kombination von Einzelzell-ATAC-Seq, das die Zugänglichkeit von Chromatin auf der Einzelzellebene abbildet, mit 3D-Chromosomenkonformationsdaten ermöglichte die Entschlüsselung von Zelltyp-spezifischen Enhancer-Promotor-Interaktionen. So konnten beispielsweise spezifische Schleifen identifiziert werden, die in myeloiden Immunzellen aktiv sind und an der Regulation von Immuncheckpoint-Genen wie CD274 (PD-L1) beteiligt sind. Dies unterstreicht die Komplexität der mikrobiologischen Regulation im Tumor und weist auf wichtige immunologische Zielstrukturen hin.
Im Rahmen der klinischen Relevanz kann das Verständnis der 3D-Genomlandschaft bei Krebs zukünftig die Entwicklung neuer diagnostischer und therapeutischer Konzepte fördern. Die Analyse von Enhancer-Rewiring verweist auf tumorarten-spezifische Biomarker und bietet Hinweise auf potenzielle Angriffspunkte für zielgerichtete Epigenom-Modifikationen. Die Erkennung von nichtkodierenden, regulatorischen Mutationen im Genom eröffnet Chancen, neue personalisierte Behandlungsstrategien zu etablieren. Besonders die Beziehung zwischen Strukturvarianten wie ecDNA und der verstärkten Aktivierung von Onkogenen kann als Grundlage für innovative Therapien dienen, etwa durch die gezielte Beeinflussung der chromatinbasierten Genregulation. Die Erforschung des dreidimensionalen Genoms bei primären menschlichen Krebserkrankungen ist allerdings noch ein junges Feld mit vielen Herausforderungen.
Die große Variabilität in der Datenqualität der Proben, die Komplexität der Tumormikroumgebung sowie die Vielzahl patientenspezifischer genetischer Veränderungen erschweren vergleichende Analysen. Dennoch zeigen die bisherigen Ergebnisse eindrucksvoll, wie wichtig die räumliche Genomorganisation für die Entstehung und Progression von Krebs ist. Die multiplen Maßstäbe – von den großen Kompartimenten über TADs bis hin zu fein abgestimmten Enhancer-Promotor-Looping-Interaktionen – bilden ein komplexes und dynamisches Regulierungsnetzwerk. Abschließend lässt sich sagen, dass die dreidimensionale Genomlandschaft eine neue Dimension für die Krebsforschung darstellt. Die Integration moderner Sequenzierungstechniken mit Chromosomenkonformationsmethoden wie HiChIP erlaubt erstmals, die molekularen Mechanismen hinter der onkogenen Genregulation in ihrem räumlichen Kontext zu verstehen.
Damit eröffnet sich ein vielversprechendes Feld für die Entwicklung neuer diagnostischer Marker und Therapien, welche die Komplexität der Genomarchitektur berücksichtigen. Die zukünftige Erforschung wird durch die Verknüpfung von 3D-Genomdaten mit klinischen Parametern und der Weiterentwicklung von Single-Zell- und Multi-Omics-Technologien weiter an Relevanz gewinnen und könnte maßgeblich zu verbesserten Behandlungsergebnissen bei Krebspatienten führen.