Die Erforschung der molekularen Zusammensetzung von Geweben ist essenziell, um biologische Prozesse und Erkrankungen umfassend zu verstehen. Dabei spielt die räumliche Verteilung verschiedener Biomoleküle wie Lipide, Metabolite, Proteine und komplexe Zuckerstrukturen eine entscheidende Rolle für physiologische Funktionen oder Pathologien. Herkömmliche Massenspektrometrie-Bildgebung (MSI) ermöglicht es, biomolekulare Verteilungen in Geweben sichtbar zu machen. Dennoch sind technische Hürden wie begrenzte räumliche Auflösung und langsame Analysezeiten bisher eine Herausforderung für die breite Anwendung. Genau an diesem Punkt setzt die innovative Methode TEMI an – Tissue-expansion Mass-spectrometry Imaging – eine Kombination aus Gewebeexpansion und hochempfindlicher MSI, welche eine Einzelzellauflösung ohne Einbußen bei der Durchsatzrate ermöglicht.
Der Kern der TEMI-Technologie liegt in einer expansiven Behandlung des Gewebes, bei der bestimmte Komponenten durch hydrogelbasierte Polymerisation chemisch verankert werden. Durch mehrfaches Einbetten in solcherart Hydrogel wird das Gewebe in mehreren Schritten vergrößert, ohne seine molekulare Integrität zu beschädigen oder wichtige Biomoleküle auszulösen. Dabei verzichtet TEMI bewusst auf aggressive Denaturierungsprozesse wie Proteinase-K-Behandlung oder proteolytische Verdauung während der Expansion, um den Verlust oder die Verlagerung von Lipiden, Peptiden, Metaboliten und Glykoproteinen zu minimieren. Stattdessen wird ein schonender Prozess unter kontrollierten Bedingungen gewählt, der eine ungefähr 2,5- bis 3,5-fache lineare Expansion ermöglicht. Die einzigartigen Vorteile von TEMI zeigen sich besonders in der räumlichen Auflösung.
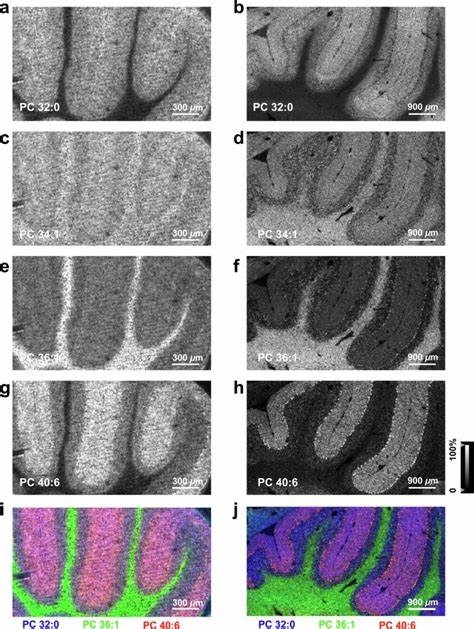
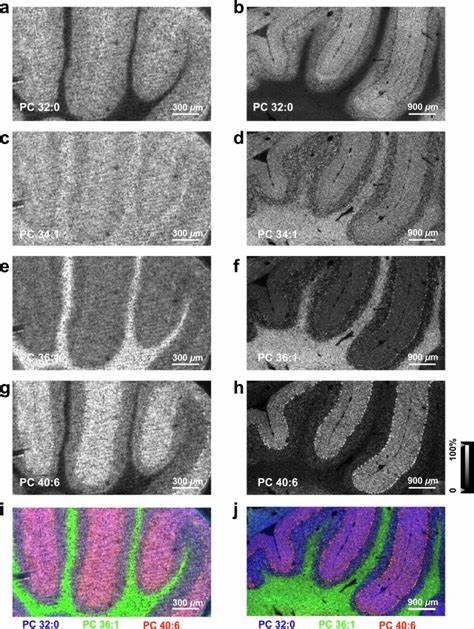
Durch die physikalische Vergrößerung des Gewebes kann die Standard-MALDI-MS-Laserablation mit 10 bis 50 Mikrometern Rastergröße angewendet werden, während die effektive Auflösung aufgrund der Expansion bei ca. 2,9 Mikrometern liegt. Dies erlaubt erstmals die Darstellung molekularer Signaturen auf Einzelzellebene, beispielsweise die spezifische Lipidverteilung in Purkinje-Zellen des Kleinhirns, die mit konventioneller MSI nur schwer erkennbar war. Diese Feinstruktur bildet die Grundlage, um funktionelle und morphologische Details auf molekularer Ebene präzise zu analysieren. Durch die Anwendung von TEMI konnten Forscher feststellen, dass verschiedene Lipidarten, Metabolite, Peptidfragmente und N-Glykane in klar voneinander abgegrenzten Arealen innerhalb komplexer Gewebe wie dem Kleinhirn oder Tumorgewebe unterschiedlich verteilt sind.
Beispielsweise zeigte TEMI die Schichtung und molekulare Differenzierung zwischen der molekularen Schicht, der Körnerschicht und der weißen Substanz im Kleinhirn sehr anschaulich. Die Lipidprofile in diesen Zonen unterschieden sich markant, was neue Erkenntnisse über die Heterogenität im Stoffwechsel und die funktionelle Spezifizierung der jeweiligen Zelltypen ermöglicht. Neben der Verbesserung der räumlichen Auflösung punktet TEMI auch in der Vielfalt der detektierbaren Moleküle. Neben Lipiden, deren Verteilung bereits gut durch herkömmliche MALDI-MSI-Methoden untersucht wird, können mit TEMI auch kleinere Metabolite, komplexe Peptide und Proteine sowie N-Glykane räumlich kartiert werden. Dies erlaubt eine multiparametrische Betrachtung des Gewebes und fördert das Verständnis biologischer Netzwerke und pathophysiologischer Veränderungen auf mehreren Ebenen.
Parallel angewandte Methoden wie immunhistochemische Anfärbung mit photocleavbaren Massentags (IHC-MALDI) ergänzen die Proteinanalyse und erhöhen die molekulare Präzision. Besonders vielversprechend ist die Anwendung von TEMI in der Krebsforschung. Tumorgewebe, das durch hohe molekulare und zelluläre Heterogenität gekennzeichnet ist, lässt sich mit TEMI besser segmentieren und analysieren. Dabei konnten bis zu 21 unterschiedliche molekulare Regionen in murinen Melanom-Modellen identifiziert werden, während klassische MSI nur wenige Regionen ohne Feindifferenzierung darstellte. Diese verbesserte Detailtiefe hat großes Potenzial in der Diagnostik, Therapieplanung und biomarkerbasierten Forschung.
Neben dem Kleinhirn und Tumorgewebe wurde TEMI erfolgreich auf weitere Organe wie Niere und Bauchspeicheldrüse angewandt. Die Methode ist somit universell einsetzbar und unterstützt die Abbildung komplexer biochemischer Landschaften in vielfältigen biologischen Systemen. Dabei bleiben geometrische Verzerrungen durch Expansion unter 12 Prozent, was die Präzision der räumlichen Analysen sichert. Der Ablauf bei TEMI beginnt mit der Fixierung des Gewebes, gefolgt von der chemischen Verankerung der Proteine durch einen AcX-Linker. Anschließend erfolgt das Einbetten in einen robusten Polyacrylamid-Hydrogel, gefolgt von weiteren Einbettungen in aufeinanderfolgenden Schritten, um die gewünschte Expansionsstufe zu erreichen.
Nach der Expansion wird das Gewebe kryoschnittfähig gemacht, und anschließend auf speziellen beschichteten Glasscheiben für die MALDI-MS-Bildgebung vorbereitet. Die Wahl der Matrix ist entscheidend für die Zielmoleküle: 1,5-Diaminonaphthalin (DAN) eignet sich optimal für Lipide, während 2,5-Dihydroxybenzoesäure (DHB) und andere Verbindungen für Peptide und Metabolite verwendet werden. Bei der Messung werden die expandierten Proben mit einem MALDI-MS-Instrument mit einem SmartBeam-Laser rasterförmig abgetastet. Dabei ist die präzise Steuerung der Laserenergie wichtig, um die Gewebeintegrität und Signalqualität zu erhalten. Die gewonnenen Spektren werden über spezialisierte Software ausgewertet, mit Fokus auf Massengenauigkeit und Signalintensität.
Die Datenanalyse umfasst auch multivariate statistische Verfahren wie bisektinge k-means Clusteranalyse zur Segmentierung molekular homogener Regionen. Natürlich bringt die Expansion eine Verringerung der lokalen Biomoleküldichte mit sich, was die Detektionssensitivität beeinflussen kann. Deshalb ist der Verzicht auf proteolytische Denaturierungsschritte so wichtig, um Verluste an Biomolekülen zu minimieren und die chemische Spezifität zu bewahren. Zukünftige Optimierungen könnten neue Verankerungsmethoden für „freie“ kleine Moleküle integrieren, um deren diffusionsbedingten Verlust während der Expansion weiter einzudämmen. Darüber hinaus ist TEMI nicht auf MALDI-MSI beschränkt.
Andere Massenspektrometrie-Imaging-Techniken wie Desorptions-Elektrospray-Ionisation (DESI) oder Laser-Ablation-Elektrospray-Ionisation (LAESI) sind kompatibel und könnten für erweiterten molekularen Kontext sorgen. Ebenso ist eine 3D-Bildgebung durch Stapelung von Kryoschnitten denkbar, wobei die Z-Auflösung vor allem von der Schnittdicke limitiert wird. Die TEMI-Technologie zeigt eindrucksvoll, wie innovative physikalisch-chemische Ansätze bestehende analytische Verfahren transformieren können. Die Kombination von Gewebeexpansion mit Massenspektrometrie-Bildgebung liefert unvergleichliche Einsichten in molekulare Landschaften biologischer Systeme und unterstützt die Entschlüsselung komplexer molekularer Muster mit relevanten Implikationen für Grundlagenforschung, klinische Diagnostik und Therapieentwicklung. Zusammenfassend bietet TEMI eine effiziente, kostengünstige und vielseitige Methode, um lipidhaltige Gewebe, metabolische Profile, proteomische Komplexität und Glykosylierungen im räumlichen Kontext mit Einzelzell-Genauigkeit zu erfassen.
Durch die schonende Behandlung wird die molekulare Integrität gewahrt und ein hohes Maß an Signalstabilität und -zuverlässigkeit erzielt. Insbesondere in der Krebsforschung, Neurowissenschaft und Organdiagnostik eröffnet TEMI neue Perspektiven. In Zukunft wird die Weiterentwicklung der Expansionstechnologien und Integration mit anderen bildgebenden Verfahren, KI-gestützter Datenanalyse und multimodalen Ansätzen die Auflösung und molekulare Tiefe weiter steigern. Die wissenschaftliche Gemeinschaft steht vor spannenden Möglichkeiten, um Gewebe mit bislang unerreichter Detailtiefe zu erkunden, was letztlich zum besseren Verständnis von Gesundheit und Krankheit beiträgt.