Die Biologie, als Wissenschaft des Lebens, offenbart eine Komplexität, die oft jenseits der Vorstellungskraft liegt. Während die Physik mithilfe mathematischer Gleichungen grundlegende Naturgesetze präzise beschreiben kann, stößt die klassische Mathematik an ihre Grenzen, wenn es darum geht, die lebendigen, sich ständig verändernden Systeme des Lebens zu verstehen. In den letzten Jahren hat jedoch das maschinelle Lernen als revolutionärer Ansatz Einzug gehalten und entpuppt sich zunehmend als die „Ursprungssprache“ der Biologie. Es bietet neue Wege, die komplexen Zusammenhänge in biologischen Systemen zu erfassen, die traditionelle Modelle nicht erreichen können. Warum ist das so und was bedeutet das für die Zukunft der biologischen Forschung und Bioingenieurwissenschaften? Diese Fragen stehen im Zentrum einer spannenden Debatte, die den Wandel vom klassischen mathematischen Paradigma hin zu datengetriebenen Modellen markiert.
Traditionelle Mathematik und ihre Einschränkungen in der Biologie Seit Jahrhunderten versuchen Wissenschaftler, die Prinzipien des Lebens mit klassischen mathematischen Methoden abzubilden. Differentialgleichungen, Wahrscheinlichkeitstheorien und statistische Modelle ermöglichen in der Physik oder Chemie eine beeindruckende Vorhersagekraft. Doch wenn biologische Systeme ins Spiel kommen, wird schnell klar, dass diese Ansätze die notwendige Detailtiefe oder Flexibilität nicht immer bereitstellen können. Die Biologie entzieht sich häufig der Simplizität und Klarheit mathematischer Formeln durch drei Hauptmerkmale: ihre enorme Dimensionalität, durchdringende Vernetzung und die Vielfalt lebender Organismen. Ein einzelner Zellorganismus besteht aus tausenden von interagierenden Genen, Proteinen und metabolischen Prozessen.
Diese Vielzahl an Elementen ist zu umfangreich für vereinfachte Gleichungen, zu klein für statistische Mittelwerte. Darüber hinaus existieren im biologischen Kontext kaum isolierte Systeme. Jedes Element beeinflusst viele andere, wodurch kontextabhängige Wechselwirkungen entstehen, die klassische Modelle selten erfassen. Schließlich lebt und entwickelt sich die Natur fortwährend, so dass starre, statische Modelle oft vom aktuellen Zustand abweichen. Aus diesem Grund erscheinen mathematische Modelle oftmals als unzureichend und limitiert, wenn es um die Beschreibung biologischer Phänomene geht.
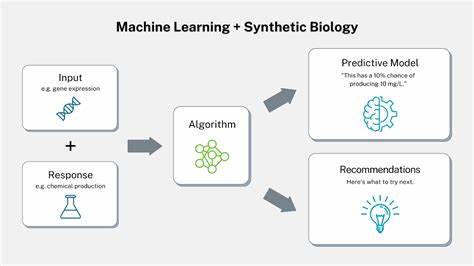
Maschinelles Lernen als neue Sprache der Biologie Im Gegensatz zu traditionellen Methoden erlaubt das maschinelle Lernen eine flexible und datengetriebene Modellierung komplexer Beziehungen. Obwohl maschinelle Lernalgorithmen mathematisch fundiert sind, unterscheiden sie sich grundlegend in ihrer Herangehensweise. Statt vom Menschen entworfene vereinfachte Modelle zu verwenden, lernen sie direkt aus riesigen Datenmengen hochdimensionale, oft nichtlineare und kontextabhängige Muster. Dies bedeutet, dass das Modell nicht versucht, die Realität im Sinne einer einfachen Gleichung zu erklären, sondern komplexe Systeme so abbildet, wie sie tatsächlich sind. Diese Fähigkeit ist besonders wertvoll, weil biologische Systeme selten lineare oder unabhängige Beziehungen zeigen.
Beispielsweise verändert der Tumorsuppressor p53 seine Wirkung radikal, abhängig vom Zelltyp oder Entwicklungsstadium. Solche Kontextabhängigkeiten lassen sich traditionell kaum mathematisch ausdrücken, während maschinelles Lernen hier glänzt und die verschiedenen Zustände gut unterscheidbar macht. Die Parallelen zwischen natürlicher Sprache und biologischer Komplexität bieten eine weitere Erklärung für die Wirksamkeit von maschinellen Lernansätzen. So wie Wörter mehrere Bedeutungen je nach Kontext annehmen, reagieren Moleküle gemeinsam auf Umweltreize, deren Interpretation sich erst durch komplexe Wechselwirkungen ergibt. Computermodelle, die ursprünglich für die Analyse von menschlicher Sprache entwickelt wurden, haben in der Bioinformatik eine neue Bedeutung erlangt und helfen, genetische Sequenzen ähnlich einem „biologischen Dialekt“ zu verstehen.
Symbolische Informationsverarbeitung in Zellen Interessanterweise spiegeln die inneren Informationsverarbeitungsprozesse von Zellen die Prinzipien des maschinellen Lernens wider. Zellen nutzen Transkriptionsfaktoren als symbolische Repräsentationen von Umweltzuständen oder inneren Signalen. Diese Proteine können in aktiven oder inaktiven Zuständen vorliegen und regulieren gezielt die Genexpression. Mit anderen Worten, sie kodieren Aufgaben und Zustände durch abstrahierte Symbole, die in Kombination hochkomplexe Antworten ermöglichen. Beispielsweise aktiviert sich beim Hitzestress ein bestimmter Transkriptionsfaktor, der dann eine Kaskade von Proteinen auslöst, welche die Zellgesundheit sichern.
Je nach Kontext verändern sich die „Bedeutungen“ dieses Faktors, vergleichbar mit dem Wort „Bank“ im Englischen. Dieses Prinzip lässt sich mit den latenten Repräsentationen in neuronalen Netzwerken vergleichen. Hohe Dimensionen biologischer Daten werden abstrahierend reduziert und bieten so eine vereinfachte, doch aussagekräftige Sicht auf komplexe Prozesse. Durch maschinelles Lernen werden genau diese latenten Dimensionen erkennbar, die biologische Zustände oder Entwicklungen wie Zellzyklusphasen oder Differenzierungsstadien repräsentieren. Dabei entstehen sogar neue Erkenntnisse, etwa wenn Clusterlösungen aus Genexpressionsdaten zeigen, dass Tumore nicht nur nach anatomischen Kriterien klassifiziert werden sollten, sondern nach molekularen Signaturen, die biologische Verwandtschaften besser beschreiben.
Die Revolution des „Predictive Biology“ Der Begriff „Predictive Biology“ steht für einen Paradigmenwechsel, der biologische Forschung grundlegend verändert. Anstatt sich auf das Beschreiben von Molekülen oder deren Wechselwirkungen zu beschränken, fragt Predictive Biology danach, wie man das Verhalten biologischer Systeme in neuen Situationen vorhersagen kann. Dieses Denken nähert sich dem Verhalten von Zellen an, die nicht die physikalischen Details begreifen müssen, sondern darauf ausgerichtet sind, Vorhersagen mit internen Signalen zu treffen, um bestmöglich auf Veränderungen zu reagieren. Im praktischen Bereich eröffnet diese Vorhersagefähigkeit ungeahnte Möglichkeiten. In der Proteinforschung etwa ermöglicht maschinelles Lernen nicht nur das Nachahmen bereits bekannter Proteinstrukturen, sondern auch die Entwicklung völlig neuartiger Proteine mit außergewöhnlichen Funktionen.
Anstelle einzelner Experimente können große Datenmengen trainiert werden, um optimale Sequenzen und Verhaltensweisen vorherzusagen und somit Entwicklungszeiten drastisch zu verkürzen. Darüber hinaus vereinfacht die Vorhersagebiologie das Verständnis und Design biologischer Systeme, die auf Vielschichtigkeit, Interaktion und Wandel basieren. Beispielsweise könnten künftig maßgeschneiderte Zelltherapien entstehen, die präzise auf die Umgebung einer Erkrankung reagieren. Ebenso sind biologische Systeme vorstellbar, die Materialien herstellen, welche bislang in Natur oder Technik unbekannt sind – all das im Einklang mit der Fähigkeit, hochkomplexe, unübersichtliche biologische Muster zu erfassen. Die unordentliche Realität der Biologie Biologie ist von Natur aus „messy“.
Selbst scheinbar klar definierte Einheiten wie Gene sind in Wirklichkeit vielschichtig und kontextabhängig, was ihre Einordnung erschwert. Ein einziges Gen kann viele unterschiedliche Proteine produzieren, je nachdem, in welchem Zelltyp es aktiviert ist oder welche Umgebungsreize vorherrschen. So zeigt das SOX9-Gen völlig unterschiedliche Funktionen – von der Geschlechtsbestimmung im Embryo bis zur Rolle in der Krebsentwicklung. Diese Variabilität macht mathematische Modelle oft untauglich, die lediglich eine einzige Funktion oder Wirkung pro Gen modellieren. Maschinenlernmodelle hingegen lernen diese Vielschichtigkeit automatisch mit ein.
Diese Fähigkeit, Vielfalt nicht als Fehler, sondern als Datenquelle zu begreifen, ist ein entscheidender Vorteil gegenüber klassischen Ansätzen. Zusätzlich provoziert die Biologie mit ihrer inhärenten Nichtlinearität und Unübersichtlichkeit ein Umdenken. Krankheit, Alterung, Entzündungen oder Stoffwechselprozesse sind keine isolierten, linear wirkenden Phänomene, sondern interagieren auf vielfache Weise. Maschinelles Lernen und datengetriebene Modelle erfassen diese Verflechtungen oft besser als bisherige Methoden. Zukunftsperspektiven für die Bioingenieurwissenschaft Bedeutet die Dominanz maschinellen Lernens in der Biologie das Ende traditioneller Ansätze? Keineswegs.
Klassische Modelle behalten gerade dort ihre Relevanz, wo sie präzise, interpretierbar und praktisch sind. Doch für die Erforschung der höchsten Komplexitätsstufen biologischer Systeme eröffnet maschinelles Lernen bisher ungeahnte Möglichkeiten. Bereits jetzt bauen bioinformatische und bioengineering-Teams auf maschinelles Lernen, um Proteinstrukturen vorherzusagen, genetische Modifikationen zu simulieren oder komplexe molekulare Netzwerke zu verstehen. Auch hybrid Modelle, die mechanistische Einsichten mit lernbasierten Methoden kombinieren, bieten vielversprechende Wege. Neural Ordinary Differential Equations sind ein Beispiel, wie sich traditionelles physikalisches Wissen mit den Vorteilen von neuronalen Netzen verbinden lässt.
Im Endeffekt stehen wir vielleicht an einem Wendepunkt der Biowissenschaften. Nachdem Jahrhunderte lang versucht wurde, die Sprache des Lebens in klassischen mathematischen Gleichungen zu formulieren, zeigen neue datengetriebene Ansätze, wie vielfältig, dynamisch und komplex biologische Systeme wirklich sind. Maschinelles Lernen bietet den Schlüssel, diese Geheimnisse zu entschlüsseln, in Mustern zu lesen und somit die Biologie in ihrer gesamten Tiefe zu begreifen. Fazit Maschinelles Lernen ist weit mehr als ein mathematisches Werkzeug; es stellt an sich eine neue Sprache dar, die besser zur vielschichtigen und vernetzten Natur biologischer Systeme passt. Durch seine Fähigkeit, Komplexität zu akzeptieren und daraus sinnvolle Vorhersagen abzuleiten, ermöglicht es tiefere Einsichten in die Funktionsweise des Lebens.
Während klassische mathematische Modelle weiterhin wichtige Funktionen erfüllen, wird maschinelles Lernen in der Biologie zunehmend zur dominanten Methodik, die Forschung, Therapie und Bioengineering revolutioniert. Das Verständnis dieser Methodik kann daher als der Schlüssel für den Fortschritt im 21. Jahrhundert gesehen werden, in dem die Verbindung von Daten, Algorithmen und biologischer Komplexität neue Horizonte öffnet.