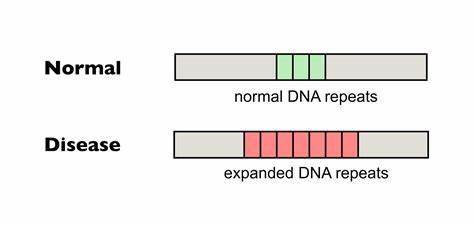
Die Huntington-Krankheit (HD) gehört zu einer Gruppe von neurologischen Erkrankungen, die durch die Expansion trinukleotidischer Wiederholungen im Genom verursacht werden. Dabei handelt es sich um eine genetische Mutation, bei der sich eine bestimmte Triplet-Sequenz, das sogenannte CAG-Repeats, im Huntingtin-Gen (HTT) vermehrt und damit toxische Effekte im Gehirn auslöst. Die Progression und Schwere der Krankheit hängt direkt von der Länge dieser CAG-Repeat-Sektionen ab, welche bei Patienten instabil sind und im Laufe ihres Lebens somatisch – also in einzelnen Körperzellen – weiter expandieren können. Dieser Dynamik wird ein zentraler Einfluss auf das Alter bei Krankheitsausbruch und den Verlauf der Huntington-Krankheit zugeschrieben. Somit bietet sich die Bearbeitung dieser Wiederholungsbereiche als ein potenzieller therapeutischer Ansatz an, der darüber hinaus noch weitere genetisch bedingte Krankheiten mit trinukleotidischen Wiederholungen betreffen könnte.
Aktuelle Forschungen setzen verstärkt auf sogenannte Basen-Editing-Technologien, eine präzise Methode der Genom-Editierung, die durch gezielte Umwandlung einzelner Nukleotide ohne Doppelstrangbrüche in der DNA den genetischen Code verändert. Besonders spannend ist dabei der Ansatz, CAG-Sequenzen in HTT mit sogenannten CAA-Interruptionssequenzen zu durchbrechen. Diese Unterbrechungen, die in der Natur bei ungefähr einem Drittel der betroffenen Personen vorkommen, etablieren stabilere Repeat-Strukturen, reduzieren die Neigung von somatischen Expansionen und verlangsamen damit die Neurodegeneration. Die synthetische Einfügung solcher Interruptionssequenzen mittels zytosinbasierender Basen-Editoren (CBEs) konnte in Zellkulturen und Tiermodellen bereits erfolgreich umgesetzt werden. Untersuchungen an Patientenfibroblasten, die eine pathologisch verlängerte CAG-Repeat-Länge aufweisen, zeigten, dass durch solche Basen-Editing-Vorgänge bis zu 82 Prozent der Zellen modifizierte HTT-Allele mit Interruptionssequenzen aufweisen.
Diese Veränderung bewirkt signifikante Verminderung der somatischen Expansionen im weiteren Zellverlauf. Während unbehandelte Zellen tendenziell eine Zunahme der CAG-Anzahl um sechs Einheiten zeigen, vermehren sich die repeat-langen Zeilen nach dem Editing nicht nur weniger, sondern zeigen sogar eine genetische Verkürzung um fünf Einheiten, was auf eine mögliche Rückbildung pathologischer Repeatlängen hindeutet. Die In-vivo-Anwendbarkeit der Methode wurde durch die erfolgreiche Nutzung adeno-assoziierter Viren (AAV) demonstriert, mit deren Hilfe ein optimierter CBE in neonatalen Mäusen mit dem Huntington-Mutationsträgermodell (Htt.Q111-Maus) ins Zentralnervensystem injiziert wurde. Die AAV9-Serotyp-Vektoren weisen eine starke Neuronotropie auf, interagieren präzise mit Hirngewebe und liefern die Editoren ohne wesentliche Nebenwirkungen.
Dies führte innerhalb weniger Wochen zu einem effizienten Einbau von Interruptionssequenzen in über 30 Prozent der HTT-Allele in Hirnrinde und Striatum. Die somatische Expansion der pathogenen CAG-Tracts reduzierte sich dadurch signifikant, und in einigen Fällen kam es sogar zu einer durch Basen-Editierung induzierten Kontraktion der Repeat-Sequenzen. Wichtig war auch die sorgfältige Überprüfung potenzieller Off-Target-Effekte. Da CAG-Repeats mehrfach im menschlichen Genom vorkommen, besteht theoretisch das Risiko, unbeabsichtigte Veränderungen an anderen Genstandorten vorzunehmen. Um die Präzision zu prüfen, wurden zahlreiche Methoden wie CIRCLE-Seq und High-Throughput-Sequenzierungen eingesetzt.
Die Ergebnisse verdeutlichen, dass der Großteil der gezielten Änderungen an nicht-codierenden oder synonymen (ungenommenen) Codons auftritt und dass potenziell schädliche Mutationen nur sehr selten und vorzugsweise an nicht-essentiellen Genloci entstanden. Zudem konnte der Einfluss von Bindungsstellen mit Mismatches zwischen Leifsequenz und Zielsequenz experimentell bewertet werden, wobei eine erhöhte Anzahl von Nukleotidabweichungen die Wahrscheinlichkeit von Off-Target-Bearbeitungen drastisch senkt. Ähnliche Erkenntnisse zur Reduktion somatischer Repeat-Expansionen wurden auch bei Friedreich-Ataxie (FRDA) gewonnen, einer weiteren Trinukleotid-Repeat-Erkrankung, deren Ursache in einer Expansion von GAA-Repeats im FXN-Gen liegt. Auch hier nutzt man Adenin-Basen-Editoren (ABEs), die gezielt A-T-Basenpaarungen in stabile G-C Paare umwandeln und damit Interruptionssequenzen in die GAA-Expansionsregion einfügen. In patienteneigenen Fibroblasten wurden damit bis zu 33 Prozent Editierungen erreicht und eine Teilrückkehr der FXN-Genexpression beobachtet, die bei FRDA maßgeblich durch die Repeat-Expansion herunterreguliert wird.
In entsprechenden Mausmodellen konnte zudem gezeigt werden, dass das Editieren der GAA-Repeats mit ABEs somatische Expansionen reduzieren und sogar die Repeat-Länge teilweise kontrahieren kann, was das therapeutische Potential dieses Ansatzes zusätzlich unterstreicht. Die Vorteile von Basen-Editing gegenüber konventionellen Methoden liegen nicht nur in der hohen Präzision und geringeren Nebenwirkungsraten, sondern auch in der Fähigkeit, bakterielle Cas9-Neckase-Funktionen bei Bedarf zu umgehen – insbesondere bei GAA-Editoren, deren Zielsequenzen im Genom zahlreich vertreten sind und bei denen potenzielle DNA-Doppelstrangbrüche die Zellintegrität gefährden könnten. Nichtsdestoweniger gibt es Herausforderungen. Zum einen sind die langfristigen Folgen der off-target Editierungen in unterschiedlichen Zelltypen und Organen noch nicht vollständig geklärt, weshalb weiterführende Studien zur Sicherheit notwendig sind. Zum anderen beschränken sich die derzeitigen AAV-Vektor-basierten Liefermethoden zumeist auf das neonatalen Gehirn, während spätere Therapiezeitpunkte und andere betroffene Organe, wie z.
B. das Herz oder die Gliazellen im Fall von FRDA, weitere Verbesserungen in der Vektorologie und Dosierung erfordern. Trotzdem markieren diese Ergebnisse einen Durchbruch in der Entwicklung genetisch basierter Therapien für Huntington und verwandte Trinukleotid-Krankheiten. Indem die Basen-Editing-Strategien Interruptionssequenzen etablieren, die die genetische Instabilität der Repeat-Tracts verringern, konnte erstmals der Schlüsselmechanismus der somatischen Expansion erfolgreich beeinflusst werden. Somit eröffnen sich neue Perspektiven für präventive und kurative Ansätze, die noch vor Überschreiten pathogener Schwellenwerte ansetzen könnten und somit den Ausbruch oder die Progression der Erkrankungen verzögern oder verhindern.
Die wissenschaftlichen Grundlagen, die aus dieser Arbeit hervorgehen, legen darüber hinaus den Grundstein für den Einsatz dieser Technologien als mächtige Werkzeuge zur Erforschung der molekularen Dynamik von trinukleotidischen Repeats und deren Interruptionsvarianten. Die klinische Anwendung basiert auf der Gewährleistung höchster Sicherheit und Effektivität, unter Berücksichtigung individueller genetischer Variationen und somatischer Mosaicismen. In Zukunft sind Innovationen bei der Vektorentwicklung, kombiniert mit zeitgemäßen Methoden der Genom-Editierung, essentiell, um eine breite und sichere Therapieumsetzung zu gewährleisten. Präzisionsmedizinische Maßnahmen, beispielsweise auf der Basis von patientenspezifischen Genomdaten und somatischer Repeat-Statistiken, könnten die Behandlungsstrategien personalisieren und optimieren. Abschließend lässt sich sagen, dass die Bearbeitung von repetitiven Trinukleotidsequenzen mittels Basen-Editing ein vielversprechendes Feld darstellt, um Huntington und andere schwere neurologische Erkrankungen zukünftig effektiver zu bekämpfen.
Die aktuellen Erkenntnisse legen einen starken Grundstein, der sowohl wissenschaftlichen als auch therapeutischen Fortschritt antreibt und neue Wege für bislang unheilbare Krankheiten ebnet.




!['Maybe Venice is the city that can save the world' [video]](/images/471FB54D-C379-4E1E-B2B4-1AC8D11C9644)