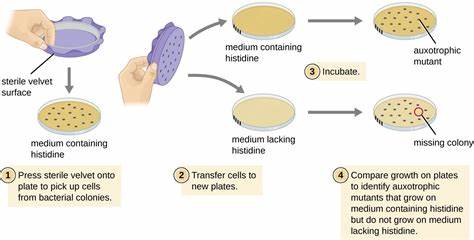
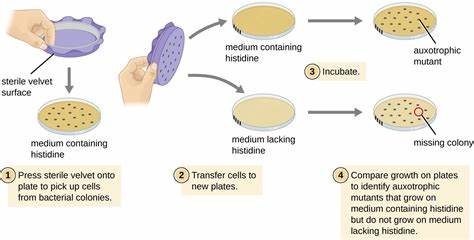
Die Identifikation genetischer Varianten, die mit menschlichen Krankheiten in Verbindung stehen, stellt eine zentrale Herausforderung in der modernen Biomedizin dar. Während die gentechnologische Revolution mit Hochdurchsatzsequenzierungen beeindruckende Datenmengen über Variationen im menschlichen Genom generiert hat, bleibt die funktionelle Analyse dieser Sequenzvarianten entscheidend für das Verständnis ihrer klinischen Relevanz. Herkömmliche Methoden wie in vitro Assays oder Untersuchungen an Zellkulturen sind zwar etabliert, jedoch häufig zeitaufwendig, kostenintensiv und in ihrer Aussagekraft begrenzt. Mit der Entwicklung von lebenden Bakterienplattformen, die menschliche Enzyme exprimieren, zeichnet sich nun ein vielversprechender neuer Ansatz ab, um die funktionelle Bedeutung genetischer Mutationen rasch und präzise abzubilden. Ein besonders innovatives Verfahren ist das sogenannte LEICA (Live E.
coli enzyme assay), bei dem das Bakterium Escherichia coli genetisch so modifiziert wird, dass wichtige bakterielle Stoffwechselenzyme durch deren menschliche Orthologe ersetzt werden. Auf diese Weise wird das Wachstum der Bakterien direkt von der Enzymaktivität des humanen Proteins abhängen, das sie heterolog exprimieren. Durch die Einführung spezifischer Mutationen, die aus Patientendaten bekannt sind, kann das Wachstumsmuster der Bakterien als funktioneller Indikator für die Wirkung dieser Mutationen auf die Enzymfunktion dienen. Diese Verbindung von mikrobieller Metabolismus und humaner Genetik eröffnet völlig neue Möglichkeiten in der Funktionsanalyse und im Medikamentenscreening. Das Prinzip beruht auf der Ähnlichkeit der Grundstoffwechselwege zwischen E.
coli und Menschen, insbesondere im Bereich der Glykolyse und des Pentosephosphatwegs. Trotz der großen evolutionären Entfernung sind viele zentrale metabolische Reaktionen konserviert, sodass bestimmte bakterielle Enzyme durch menschliche Varianten ersetzt werden können, ohne dass der Metabolismus vollständig zusammenbricht. Im LEICA-System wurden beispielsweise die bakteriellen Enzyme Phosphoglucoseisomerase (pgi) und Glucose-6-phosphatdehydrogenase (zwf) durch entsprechende menschliche Enzyme ersetzt. Das Wachstum der modifizierten Bakterien wird dadurch von der Aktivität des humanen Enzyms bestimmt und reflektiert so dessen funktionelle Effizienz. Die praktische Umsetzung umfasst die präzise genetische Erzeugung von E.
coli-Stämmen, bei denen die bakteriellen Gene für die Zielenzyme deletiert und durch menschliche Genkonstrukte ergänzt werden. Darüber hinaus können beliebige Mutationen in den humanen Enzymgenen gezielt eingebracht werden, um ihre funktionellen Folgen zu analysieren. Die Wachstumsgeschwindigkeit der Bakterien in definierten Medien wird dabei quantitativ gemessen und mit enzymatischen Aktivitäten in vitro sowie klinischen Daten korreliert. Studien zeigen eine beeindruckende Übereinstimmung, die das LEICA-System als zuverlässiges und schnelles Werkzeug zur funktionellen Charakterisierung menschlicher Mutationen etabliert. Ein besonders interessantes Anwendungsfeld ist die Untersuchung von erblichen enzymopathischen Erkrankungen.
So konnte etwa der Einfluss bekannter GPI-Mutationen, die mit hämolytischer Anämie assoziiert sind, im LEICA-System nachvollzogen werden. Pathogene Mutationen führten zu deutlich verringerter bakterieller Wachstumsrate, während benigne Varianten kaum einen Effekt zeigten. Ebenso wurden zahlreiche Varianten des G6PD-Enzyms, dem häufigsten Enzymdefekt weltweit, funktionell bewertet und Klassifizierungen bezüglich ihrer Pathogenität unterstützt. Bemerkenswert ist die Möglichkeit, auch Varianten unbekannter klinischer Relevanz schnell einzuordnen und so neue Erkenntnisse zum Krankheitsgeschehen beizusteuern. Über die reine Charakterisierung genetischer Mutationen hinaus eröffnet LEICA auch vielversprechende Perspektiven für das Screening von Medikamenten und Wirkstoffkandidaten.
Durch Zugabe kleiner Moleküle in das Bakterienwachstumsmedium lassen sich Inhibitoren oder Aktivatoren der humanisierten Enzyme direkt auf ihre Wirksamkeit testen. Ein Beispiel ist die Anwendung des G6PD-Aktivators AG1, der über LEICA seine enzymaktivierende Wirkung in lebenden Zellen bestätigte. Ebenso konnten mehrere bekannte und neuartige G6PD-Inhibitoren aus einer Wirkstoffbibliothek herausgefiltert werden, was LEICA zu einer kostengünstigen und hochdurchsatzfähigen Plattform für die Wirkstoffsuche macht. Der Einsatz von LEICA bringt zudem Vorteile gegenüber traditionellen Methoden mit. Im Gegensatz zu aufwändigen und teuren In-vitro-Assays erfordert es keine isolierten Proteine und aufwendige biochemische Analysen.
Im Vergleich zu eukaryotischen Zellkulturen ist E. coli leicht genetisch manipulierbar, wächst schnell und ist perfekt für großskalige Tests geeignet. Die natürliche Anwesenheit von Substraten, Cofaktoren und zellulären Komponenten schafft darüber hinaus eine aus der Physiologie abgeleitete Umgebung, die In-vitro-Bedingungen oft fehlt. Diese Kombination macht LEICA besonders attraktiv für die schnelle Validierung zahlreicher genetischer Varianten, die in der Bevölkerung häufig vorkommen. Nicht zuletzt bietet diese Methode wichtige Unterstützung für die Präzisionsmedizin.
Da individuelle genetische Profile oft zahlreiche Varianten enthalten, deren Funktion unklar ist, ist die Fähigkeit zur schnellen funktionellen Bewertung eine Grundvoraussetzung für personalisierte Therapieansätze. LEICA kann hier helfen, Mutationen hinsichtlich ihrer pathogenen Bedeutung rasch zu unterscheiden und potenzielle medikamentöse Interventionen zu testen, was gerade bei enzymatischen Störungen von hohem klinischem Nutzen ist. Obwohl LEICA vielversprechend ist, sollten auch Limitationen beachtet werden. Das bakterielle System ist ein Modell, das menschliche Enzyme in einem prokaryotischen Kontext exprimiert. Unterschiede in Posttranslationen, Proteininteraktionen oder Zellkompartimentierung, die in humanen Zellen existieren, können nicht vollständig abgebildet werden.
Des Weiteren kann die bakterielle Membranpermeabilität und Aufnahme von Wirkstoffen unterschiedlich sein, was die Übertragung von Ergebnissen auf humane Zellen beeinflussen kann. Trotzdem wurden in Studien geeignete Kontrollmaßnahmen implementiert, um Fehlinterpretationen möglichst zu vermeiden, etwa die Gegenprüfung mit E. coli-Stämmen ohne humanisierte Enzyme. Zukünftige Weiterentwicklungen können die Bandbreite der getesteten Enzyme deutlich erweitern und weitere molekulare Mechanismen integrieren. Die Kombination mit automatisierter Genomeditierung und Hochdurchsatz-Wachstumsmonitoring erlaubt zudem eine Beschleunigung und Skalierung des Prozesses.
Innovative Schnittstellen mit computergestützten Analysemodellen könnten zudem die Interpretation der erhobenen Daten verbessern und die Vorhersagekraft klinischer Folgen von Mutationen stärken. Insgesamt markiert die Implementierung lebender Bakterien als funktionelle Assay-Plattform einen bedeutsamen Fortschritt in der Erforschung genetischer Erkrankungen und der Arzneimittelentwicklung. LEICA vereint die Vorteile präziser genetischer Manipulation, biologischer Aussagekraft und Operationalisierung in einem System, das sowohl Forscher als auch Klinikern neue Einblicke bietet. Es ermöglicht eine schnelle, unkomplizierte und kosteneffiziente Evaluierung menschlicher Enzymvarianten, unterstützt die Suche nach therapeutischen Substanzen und fördert letztlich die Umsetzung von Erkenntnissen in eine gezielte, personalisierte Behandlung. Die Integration solcher innovativen biotechnologischen Plattformen wird die Zukunft der humanen Genetik und Biomedizin maßgeblich prägen.
LEICA stellt dabei einen vielversprechenden Baustein dar, der das Potenzial besitzt, die Diagnose, das Verständnis und die Behandlung genetischer Erkrankungen nachhaltig zu verbessern und damit einen wichtigen Beitrag zur Weiterentwicklung der Präzisionsmedizin zu leisten.