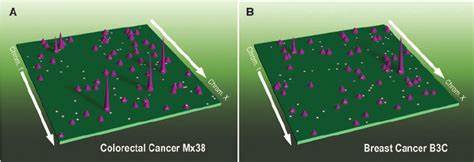
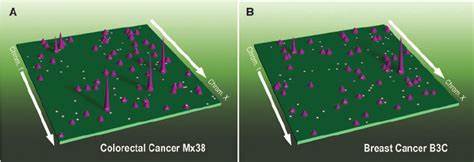
Das menschliche Genom ist nicht nur eine lineare Abfolge von DNA-Basen, sondern ein komplex gefaltetes System, das in einem winzigen Zellkern von etwa 10 Mikrometern Durchmesser untergebracht ist. Diese dreidimensionale Organisation ermöglicht es, dass entfernte regulatorische Elemente, sogenannte Enhancer, mit Promotoren korrespondieren und so die spezifische Genaktivität steuern. Insbesondere bei Krebs, einer Erkrankung, die durch genetische Veränderungen und epigenetische Dysregulation charakterisiert ist, gewinnt die räumliche Genomanordnung zunehmend an Bedeutung für das Verständnis der Krankheitsqualität und ihrer therapeutischen Angreifbarkeit. Forschungen an 69 Tumorproben aus 15 verschiedenen primären menschlichen Krebsarten im Rahmen von Studien des Cancer Genome Atlas bieten faszinierende Einblicke in diese 3D-Genomlandschaft. Mithilfe der innovativen HiChIP-Technologie, die die Aktivität von Enhancern und deren physische Verbindungen zu Promotoren in hoher Auflösung abbildet, konnten über 665.
000 signifikante Interaktionen identifiziert werden. Diese sogenanntes Enhancer-Promotor-Netzwerk ist von zentraler Bedeutung für die Aktivierung von Genen, die für die Tumorentstehung und das Fortschreiten relevant sind. Eins der grundlegenden Ergebnisse dieser Untersuchung ist die Erkenntnis, dass es drei Archetypen der Enhancer-Nutzung bei über 100 bekannten Onkogenen gibt. Einige Gene nutzen statische Enhancer, die konsistent aktiv sind, andere zeigen selektive Gain-Muster—also eine verstärkte Aktivierung nur in bestimmten Krebsarten—und wieder andere präsentieren dynamische Neuanordnungen ihres Enhancer-Netzwerks. Dieses differenzierte Bild illustriert, wie flexibel und anpassungsfähig die Genregulation in Tumoren sein kann und weist auf die Bedeutung der 3D-Genomorganisation für die Erkennung und Deutung von therapeutischen Zielstrukturen hin.
Die Analyse geht jedoch weit über die bloße Kartierung von Enhancer-Promotor-Interaktionen hinaus. Sie integriert auch Daten von tiefgehender Ganzgenomsequenzierung, um strukturelle Varianten zu identifizieren, die die Genomarchitektur verändern können. Solche strukturellen Änderungen, darunter Inversionen, Translokationen und fokale Amplifikationen, können die Positionen von Enhancern und Promotoren im dreidimensionalen Raum verändern und so zu einem sogenannten „Enhancer-Rewiring“ führen. Dieser Prozess kann Onkogene in neuen Konstellationen mit stärkenden regulatorischen Elementen verbinden, was ihre Überexpression fördert und somit das Wachstum und die Aggressivität von Krebszellen unterstützt. Ein besonders bemerkenswertes Phänomen ist die Rolle von extrachromosomaler DNA (ecDNA), die als zirkuläre DNA-Elemente außerhalb der chromosomalen DNA existiert.
Sie ermöglicht eine besonders intensive Verstärkung von Onkogenen und zeigt auch umfangreiches Enhancer-Rewiring. Im Vergleich zu anderen Arten von strukturellen Veränderungen schaffen ecDNA-Amplifikationen viele neue Enhancer-Promotor-Schleifen und können so eine massive gesteigerte Genaktivität erzeugen, die mit schlechten Prognosen bei Patienten verbunden ist. Dieser Befund hebt ecDNA als wichtigen Akteur in der Tumorbiologie hervor und bietet spannende neue Ansatzpunkte für therapeutische Interventionen. Zusätzlich zur Tumorzelle selbst besteht der Tumormikroumgebung, also die Gesamtheit der Zellen und Gewebe um den Tumor herum, aus einer Vielzahl von nichttumorösen Zelltypen, darunter Immunelemente wie Myeloide, T- und B-Zellen. Die Erforschung der 3D-Genomstruktur unter Einbezug von Einzellsequenzierungsdaten zeigt, dass auch in diesen Umgebungszellen enhancer-vermittelte Regulationen vorhanden sind, die wichtige Gene, beispielsweise Immune-Checkpoints wie PD-L1, steuern.
So konnte identifiziert werden, dass gewisse Enhancer-Promotor-Interaktionen spezifisch für Myeloidzellen sind, was das Verständnis für die Immunmodulation im Tumormikroumfeld erweitert und neue Einblicke in die Mechanismen der Immunflucht von Tumoren bietet. Parallel zur Entdeckung der räumlichen Chromatinstruktur wurden auch nichtkodierende Mutationen näher betrachtet, die in regulatorischen Elementen auftreten. Diese Mutationen können neue Bindungsstellen für Transkriptionsfaktoren schaffen oder bestehende verändern und so die Aktivität von Enhancern steigern. Ihre Identifikation ist deshalb enorm wichtig, da sie zur Aktivierung von Onkogenen beitragen können, selbst wenn sie nicht direkt in den kodierenden Bereichen der DNA stattfinden. Zum Beispiel wurde eine Mutationen in einem Enhancer des FGFR1-Gens bei Blasenkrebs gefunden, die eine starke Verstärkung der Enhanceraktivität und folglich der Genexpression bewirkt und mit einem schlechten klinischen Verlauf korreliert.
Die HiChIP-Technik bietet ferner die Möglichkeit, strukturelle Varianten nicht nur durch klassische genomische Sequenzierung zu entdecken, sondern auch ihre Auswirkungen auf die 3D-Architektur in der Zelle zu visualisieren. Die Verbindung von genomischem Breakpoints mit der Formation sogenannter ‚Neoloops‘, also neuen chromosomalen Schleifen, verdeutlicht, wie strukturelle Variationen die chromatinbasierte Genregulation reorganisieren. Translokationen zeigen hierbei im Vergleich zu anderen strukturellen Veränderungen hohes Potenzial für das Schaffen vieler neuer E–P-Verknüpfungen. Dies unterstreicht die Komplexität des genomischen Repertoires, mit denen Tumorzellen die Genregulation effizient umgestalten. Diese systematische Kartierung und Integration von 3D-Genomdaten mit genomischen und epigenetischen Informationen bietet eine reichhaltige Ressource zum besseren Verständnis der Genregulationsmechanismen in Krebs.
Die Erkenntnis, dass die Gesamtstruktur von Chromatinkompartimenten und TADs (topologisch assoziierte Domänen) zwar relativ stabil bleibt, die feine Regulierung durch Enhancer-Promotor-Schleifen jedoch hochdynamisch und krebsartspezifisch ist, verändert die Perspektive auf Erbgutveränderungen und deren funktionale Bedeutung. Darüber hinaus haben diese Studien auch praktische Implikationen in der klinischen Onkologie. Das Hervorheben, welche Onkogene hauptsächlich durch DNA-Amplifikationen gesteuert werden und welche vor allem auf enhancer-vermittelte Regulation zurückzuführen sind, kann helfen, Patientengruppen zu definieren, die von unterschiedlichen Therapiestrategien profitieren könnten. So könnten beispielsweise Patienten mit Onkogenamplifikationen eventuell besser auf Therapien ansprechen, die die Genomkopienzahl adressieren, während andere von epigenetischen oder regulatorischen Therapien profitieren könnten. Neben dem tiefen Verständnis der molecularen Mechanismen öffnet die Erforschung der 3D-Genomlandschaft auch den Blick auf neue Therapeutika.