Die Analyse der molekularen Zusammensetzung von Geweben hat sich in den letzten Jahren zu einem zentralen Bestandteil moderner biomedizinischer Forschung entwickelt. Insbesondere das Verständnis der räumlichen Verteilung von Biomolekülen wie Lipiden, Metaboliten, Proteinen oder Glykanen trägt wesentlich dazu bei, biologische Prozesse im Organismus besser zu verstehen und Krankheiten gezielter zu diagnostizieren und zu behandeln. Die Tissue-Expansion Mass-Spectrometry Imaging, kurz TEMI, bietet in diesem Zusammenhang eine bahnbrechende Methode, die sowohl eine erhöhte räumliche Auflösung als auch eine tiefgehende molekulare Analyse von Gewebeproben ermöglicht. TEMI kombiniert gezielte Gewebeexpansion mit massenspektrometrischer Bildgebung und hebt somit die Grenzen bisheriger Technologien auf eine neue Ebene. Die Herausforderung bei der molekularen Bildgebung von Geweben liegt darin, biomolekulare Informationen mit hoher chemischer Spezifität und zugleich mit einer feinen räumlichen Auflösung abzubilden.
Klassische Massenspektrometrie-Methoden erfahren häufig Einschränkungen durch eine begrenzte räumliche Auflösung, wodurch molekulare Prozesse auf Einzelzellebene oder innerhalb komplexer Gewebestrukturen nur unzureichend erfasst werden können. Hochauflösende Massenspektrometrie-Instrumente sind oft mit hohen Kosten und komplexer Handhabung verbunden, was ihre Verbreitung behindert. TEMI adressiert genau diese Probleme durch eine innovative Kombination aus physikalischer Gewebeexpansion und etablierter MALDI-Massenspektrometrie (Matrix Assisted Laser Desorption Ionization). Durch die Vergrößerung des Gewebes wird die vorhandene räumliche Struktur physisch aufgeweitet, sodass herkömmliche Laserstrahlgrößen in der Massenspektrometrie eine deutlich feinere Detailauflösung erzielen können, ohne aufwendige Modifikationen am Instrument selbst. Das Grundprinzip von TEMI beruht auf einem mehrstufigen Verfahrensablauf, bei dem biologische Gewebe zunächst durch die Einbettung in ein hydrogelartiges Polymernetzwerk chemisch stabilisiert und fixiert werden.
Im Gegensatz zu traditionellen Expansionsverfahren, die oftmals Proteolyse oder aggressive Denaturierungsschritte vorsehen, verzichtet TEMI bewusst auf solche Behandlungsschritte, um die native molekulare Anordnung und insbesondere nicht-kovalent gebundene Moleküle wie Lipide oder Metabolite bestmöglich zu erhalten. Stattdessen erfolgt die Expansion kontrolliert in mehreren Runden, wobei jeder Schritt zu einer homogen isotropen Ausdehnung des Gewebes führt. Durch diese Anpassung wird sichergestellt, dass Strukturen bis in den Mikrometerbereich präzise abgebildet werden können, ohne dabei die chemische Integrität der Moleküle zu beeinträchtigen. Die Umsetzung von TEMI erfordert auch eine Anpassung der Probenvorbereitung, insbesondere bei der Kryoschnitte-Technik. Hier wurde eine optimale Verwendung von Saccharoselösungen wie einer 30-prozentigen Saccharose im PBS-Puffer entwickelt, um die Verarbeitbarkeit der expandierten Gewebeabschnitte bei etwa 30 Mikrometer Schnittdicke zu gewährleisten.
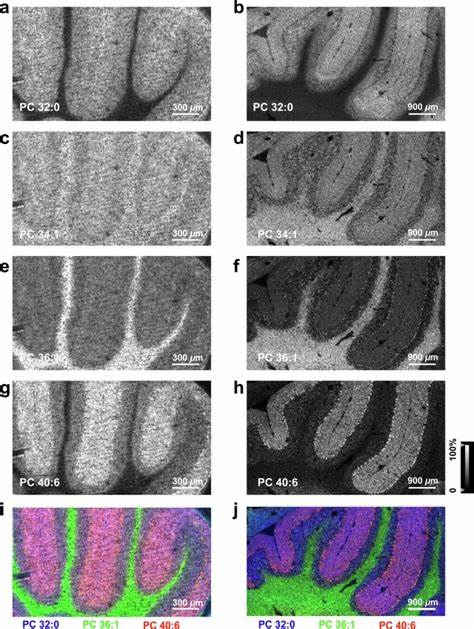
Dies stellt sicher, dass sowohl die physische Struktur als auch die molekularen Bestandteile intakt bleiben und anschließend einer MALDI-Massenspektrometrie zugeführt werden können. Die Auswahl der Matrix-Materialien wurde ebenfalls optimiert, um eine effiziente Ionisierung unterschiedlichster Biomoleküle im expandierten Gewebe zu ermöglichen. Durch die physikalische Expansion kombiniert mit einem geeigneten Rasterlaser im MALDI-Prozess lassen sich Bildgebungsauflösungen erreichen, die eine effektive Pixelgröße von bis zu 2,9 Mikrometern aufweisen. So können beispielsweise in Mäusegehirnen einzelne Purkinje-Zellen gezielt visualisiert und ihre lipidomische Zusammensetzung analysiert werden – ein bislang nur mit sehr komplexen und teuren Instrumenten realisierbarer Wert. Darüber hinaus zeigt TEMI eine hohe Sensitivität für eine Vielzahl biomolekularer Klassen, darunter Phosphatidylcholine, Glykolipide, Peptide sowie N-Glykane, und kann somit ein umfassendes Bild von komplexen molekularen Landschaften in Geweben liefern.
Ein besonderer Vorteil von TEMI besteht darin, dass es sich problemlos auf verschiedenste Organ- und Gewebetypen anwenden lässt. Neben dem Gehirn konnten beispielsweise Nieren, Bauchspeicheldrüse und auch Tumorgewebe von Mäusen erfolgreich expandiert und mit hoher molekularer Auflösung analysiert werden. Insbesondere in Tumorgeweben offenbart TEMI eine beachtliche metabolische Heterogenität, die herkömmlichen Massenspektrometrieverfahren verschlossen bleibt. Mittels spektraler Clusteranalysen lassen sich Tumorregionen mit unterschiedlichen molekularen Profilen segmentieren, was nicht nur eine bessere Charakterisierung der Tumormikroumgebung ermöglicht, sondern auch wichtige Erkenntnisse für personalisierte Therapien liefern kann. Die Kompatibilität von TEMI mit gängigen MALDI-MSI-Systemen macht die Methode besonders attraktiv für Forschungslabore, da keine teuren Spezialgeräte notwendig sind.
Darüber hinaus ist TEMI vielfältig anpassbar und lässt sich theoretisch auch mit anderen Ionisationsmethoden, wie der Desorptions-Elektrospray-Ionisation (DESI) oder der Laserablation zum Beispiel, verbinden. Ein weiterer Pluspunkt ist die Kompatibilität mit korrelativer Bildgebung: fluoreszenzmikroskopische Verfahren können parallel angewendet werden, um strukturelle und molekulare Daten innerhalb des gleichen probenden Gewebes zu integrativ zu analysieren. Trotz der überzeugenden Vorteile gilt es, einige technische Herausforderungen zu beachten. Die ausgedehnte Gewebeexpansion führt zwangsläufig zu einer Verdünnung der Biomoleküle, was die absolute Signalintensität in der Massenspektrometrie etwas schwächt. Aus diesem Grund sind Optimierungen der Probenvorbereitung, beispielsweise hinsichtlich Schnittdicke und Matrixauftrag, notwendig, um ein optimales Gleichgewicht zwischen räumlicher Auflösung und Nachweisgrenze zu erreichen.
Zudem können frei gelöste kleine Moleküle, besonders wasserlösliche Metabolite, bei der Expansion diffundieren oder verloren gehen. Hier sind zukünftige Forschungsarbeiten gefragt, um die chemische Verankerung oder Fixierung solch kleiner Moleküle weiter zu verstärken und eine verlustfreie Abbildung sicherzustellen. Neben den rein analytischen Anwendungen bietet TEMI auch vielversprechende Möglichkeiten für die klinische Forschung. Die detaillierte molekulare Kartierung von Tumoren eröffnet neue Perspektiven zur Aufdeckung intratumoraler Heterogenität, was für die Entwicklung präziser Diagnostik und maßgeschneiderter Therapiestrategien von zentraler Bedeutung ist. Auch im Bereich der Neurowissenschaften kann TEMI dabei helfen, molekulare Besonderheiten in gesundem sowie erkranktem Hirngewebe aufzulösen und somit die Pathophysiologie neurodegenerativer Erkrankungen besser zu verstehen.
Angesichts der vergleichsweise einfachen Implementierung und der umfassenden Informationsgewinnung wird TEMI voraussichtlich in den nächsten Jahren zu einem wichtigen Werkzeug sowohl in der Grundlagenforschung als auch in der biomedizinischen Diagnostik avancieren. Die Kombination aus hoher räumlicher Präzision, multiparametrischer molekularer Analyse und breiter Anwendbarkeit weist TEMI als eine Schlüsseltechnologie für die nächste Generation der molekularen Gewebeabbildung aus. Zusammenfassend stellt Tissue-Expansion Mass-Spectrometry Imaging eine bahnbrechende Innovation dar, die physikalische Gewebeexpansion mit moderner Massenspektrometrie effizient verknüpft. Sie ermöglicht eine molekulare Bildgebung mit Einzelzellauflösung, ohne aufwändige Geräteupgrades oder kostspielige Technik. TEMI deckt ein breites Spektrum biomolekularer Klassen ab und eignet sich für verschiedenste Gewebetypen und Fragestellungen.
Damit eröffnet TEMI neue Horizonte für die Erforschung komplexer biologischer Systeme, die Entschlüsselung pathologischer Mechanismen und die Entwicklung innovativer therapeutischer Ansätze.