Die rasante Entwicklung der Künstlichen Intelligenz (KI) hat längst auch im Bereich der Biowissenschaften zu revolutionären Fortschritten geführt. Insbesondere die Fähigkeit, Proteine gezielt zu entwerfen und zu manipulieren, wird zunehmend durch KI-gestützte Technologien ermöglicht. In einem spannenden Experiment stellte ich einer KI die Aufgabe, ein Protein zu kreieren. Das Ergebnis zeigt nicht nur das Potenzial dieser neuen Methoden, sondern wirft auch interessante Fragen zu Chancen und Limitationen der Technologie auf. Proteine sind die Arbeitspferde des Lebens.
Sie erfüllen zahlreiche Funktionen in Organismen, von der Strukturgebung über den Stoffwechsel bis hin zur Immunabwehr. Ihre komplexe Faltung und hochspezifische Sequenz aus Aminosäuren machen es schwierig, neue Proteine mit gewünschten Eigenschaften manuell zu entwickeln. Hier setzt KI an. Durch das Training auf enorm großen Datensätzen von bekannten Proteinsequenzen und Strukturen kann eine KI Muster erkennen und daraus neue, bisher unentdeckte Sequenzen generieren. Mein Experiment begann mit der Nutzung eines sogenannten Protein Language Models, einer KI, die ähnlich wie Sprachmodelle funktioniert, aber statt Wörter Aminosäuresequenzen generiert.
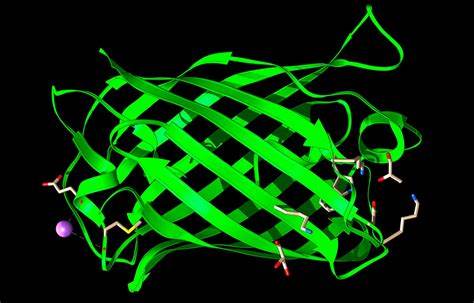
Nach wenigen Eingaben erhielt ich eine von der KI geschaffene Sequenz, die den Grundbaustein eines Proteins darstellt. Natürlich war die erste Kreation noch fern von einem funktionellen Biomolekül, eher ein „Rohentwurf“. Doch die Möglichkeit, innerhalb kürzester Zeit völlig neue Sequenzen zu erzeugen, ist beeindruckend. Die zugrundeliegenden KI-Modelle profitieren von enormen Datenmengen. Ein konkretes Beispiel ist das Pinal-Modell, das auf 1,7 Milliarden Paaren aus Proteinsequenzen und Textbeschreibungen trainiert wurde.
Dadurch versteht die KI nicht nur die rein biochemische Sprache, sondern auch den funktionellen Kontext. Dies erlaubt es Forschern, Proteine gezielt für bestimmte Zwecke zu designen, etwa Enzyme mit höherer Effizienz oder therapeutische Moleküle mit besonderer Stabilität. Neben der reinen Sequenzgenerierung helfen KI-Systeme bei der Vorhersage der Proteinstruktur – ein entscheidender Schritt zu einem funktionierenden Protein. Erst die räumliche Faltung bestimmt, ob und wie ein Protein seine Aufgabe erfüllen kann. Dank Deep-Learning-Modellen wie AlphaFold wurde dieses komplexe Problem der Faltung zunehmend lösbar, was einen Meilenstein in der Strukturbiologie darstellt.
Die Verbindung dieser beiden KI-Fähigkeiten – Sequenzdesign und Strukturvorhersage – verspricht enorme Fortschritte für die Biotechnologie. Hersteller können maßgeschneiderte Proteine entwerfen, die beispielsweise Medikamente verbessern, industrielle Prozesse effizienter machen oder Umweltschutztechnologien ermöglichen. Dies eröffnet neue Wege in der personalisierten Medizin, indem man individuelle Therapien mit speziell angepassten Proteinen entwickelt. Doch die Arbeit mit KI-generierten Proteinen steht noch am Anfang. Viele der entworfenen Sequenzen sind theoretischer Natur und müssen erst im Labor synthetisiert und getestet werden.
Die biologische Funktionalität, Stabilität im Körper oder mögliche immunologische Reaktionen sind kritische Faktoren, die experimentell validiert werden müssen. Hier trifft die High-Tech-Welt der Computermodelle auf die Realität der Biochemie. Ethik und Sicherheit spielen ebenfalls eine bedeutende Rolle. Der freie Zugang zu KI-Tools und großen genetischen Datenbanken erfordert verantwortungsvollen Umgang. Missbrauch könnte Risiken bergen, etwa bei der Entwicklung schädlicher biologischer Agenzien.
Daher setzen Forschungseinrichtungen und Regulierungsbehörden verstärkt auf Richtlinien und transparente Verfahren, um Innovation und Sicherheit in Einklang zu bringen. Inzwischen gibt es zahlreiche akademische und kommerzielle Initiativen, die KI für die Proteinentwicklung einsetzen. Oftmals sind diese Angebote über Webserver frei zugänglich, was die Forschung demokratisiert und Kollaborationen erleichtert. Gleichzeitig entwickeln Entwickler Open-Source-Modelle, um die Community weiter zu stärken. Ein weiterer spannender Aspekt ist die Integration von multimodalen Daten.
Neben Sequenzen und Strukturen können KI-Modelle künftig auch Verhaltensdaten, metabolische Informationen und Umgebungsfaktoren einbeziehen. Durch diese Vernetzung wird die Proteinentwicklung noch präziser und auf spezifische Anwendungen zugeschnitten. Mein persönliches Experiment zeigt eindrücklich, wie intuitiv und zugänglich moderne KI-Technologien geworden sind. Mit wenigen Zeilen Code lassen sich Kreativprozesse starten, die früher wochenlange manuelle Arbeit erfordert hätten. Zugleich erinnert es daran, dass Wissenschaft immer ein Zusammenspiel aus Computermodellen, Laborexperimenten und kritischer Analyse bleibt.
Die Zukunft der Proteinentwicklung mit KI birgt enormes Potenzial. Insbesondere die schnelle Generierung neuer Moleküle kann Therapien revolutionieren, nachhaltige Bioprodukte hervorbringen und das Verständnis des Lebens auf molekularer Ebene vertiefen. Dennoch ist es wichtig, den Fortschritt verantwortungsvoll zu begleiten – mit Transparenz, ethischer Reflexion und interdisziplinärer Zusammenarbeit. Zusammenfassend lässt sich sagen, dass KI die Biologie grundlegend verändert. Von der Entdeckung bis zur Anwendung schafft sie Werkzeuge, die menschliches Wissen erweitern und neue Horizonte öffnen.
Mein Experiment ist nur ein kleiner Blick in diese aufregende Welt, die in den kommenden Jahren noch viele weitere Durchbrüche bereithalten wird.