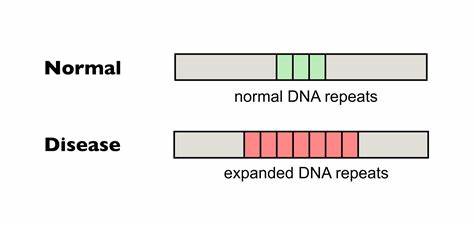
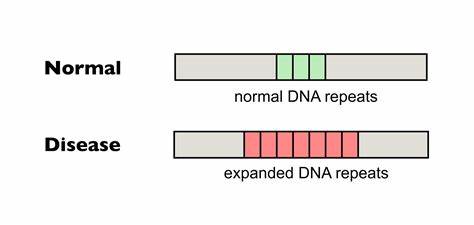
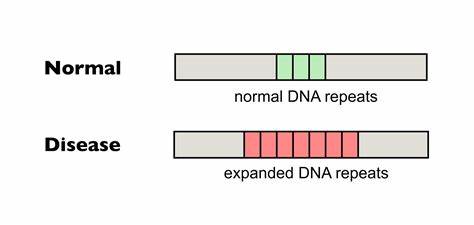
Huntington-Krankheit (HD) gehört zu den bekanntesten neurodegenerativen Erkrankungen, deren molekulare Ursache auf einer genetischen Besonderheit beruht: der Expansion von CAG-Trinukleotid-Wiederholungen im Huntingtin-Gen (HTT). Diese Wiederholungen führen zu pathologischen Veränderungen, die eine progressive Degeneration des Nervensystems nach sich ziehen. Ein vielversprechender Forschungsansatz beschäftigt sich aktuell mit der gezielten Bearbeitung dieser DNA-Wiederholungen, um somatische Expansionen im Genom der Patientenzellen zu verringern und somit den Krankheitsverlauf zu beeinflussen. Die genetische Grundlage von Huntington besteht im Anstieg der Anzahl von CAG-Trinukleotid-Wiederholungen, die kodieren das Aminosäure-Glutamin. Längere Wiederholungsstränge korrelieren direkt mit einem früheren Krankheitsbeginn und einem schwereren Verlauf.
Doch nicht nur die Länge der Wiederholungen zum Zeitpunkt der Geburt ist entscheidend, sondern auch deren Instabilität im Laufe des Lebens. Somatische Expansionen der CAG-Repeat-Sequenzen in verschiedenen Zelltypen, insbesondere in Neuronen, tragen zur Progression der Erkrankung bei. Dabei bilden sich atypische DNA-Strukturen, sogenannte Schleifen oder R-Loops, die die DNA-Reparaturmechanismen des Körpers fehldirektionieren und zu weiteren Verlängerungen der CAG-Repeats führen. Bislang konnte keine zugelassene Therapie die somatische Expansion direkt beeinflussen oder stoppen. Die Forschung sucht daher nach innovativen Wegen, die genetische Ursache zu adressieren und so die neurodegenerative Progression zu bremsen oder gar zu verhindern.
Die Verwendung von präzisen Genom-Editierungstechniken, insbesondere sogenannter Base-Editoren, tritt hierbei zunehmend in den Vordergrund. Diese Technologie erlaubt es, gezielte Basenaustausche in der DNA vorzunehmen, ohne dass dabei Doppelstrangbrüche entstehen, wodurch die Gefahr für unerwünschte Folgen minimiert wird. Aktuelle Studien aus dem Jahr 2025 zeigen, dass durch die gezielte Einführung von einzelnen Basen innerhalb der CAG-Repeats sogenannte „Interruptions“ geschaffen werden können. Diese Interruptions, meist in Form von CAA-Codons, sind natürliche Varianten eines gesunden Allels und stabilisieren die Wiederholungsregion. Ihre Anwesenheit reduziert signifikant die Wahrscheinlichkeit und das Ausmaß somatischer Expansionen.
Im Gegensatz zu den pathologischen, ununterbrochenen CAG-Strängen verhindern diese Interruptions die Bildung von DNA-Schleifen und die damit verbundenen fehleranfälligen Reparaturprozesse. Das Verfahren beruht darauf, Cytosin-Base-Editoren (CBEs) gezielt auf die CTG-Sequenz auf der gegensätzlichen DNA-Strangseite auszurichten. Durch molekulare Werkzeuge, die an spezifische DNA-Sequenzen binden, wird Cytosin (C) in Thymin (T) umgewandelt, was im Endeffekt zu einer Umwandlung von CAG zu CAA auf dem codierenden Strang führt. Diese kleinen Veränderungen sind synonym und beeinflussen die Aminosäuresequenz des Huntingtin-Proteins nicht, verhindern jedoch wirksam die instabile Expansion der Repeat-Sequenzen. Experimentelle Arbeiten in Patientenzellen und Tiermodellen untermauern die Wirksamkeit dieses Ansatzes.
In humanen Fibroblasten von HD-Patienten konnte nach Behandlung mit CBEs eine signifikante Reduktion somatischer Repeat-Expansionen beobachtet werden. Ebenso wurden in transgenen Mäusen des Htt.Q111 Modells nach Verabreichung von AAV9-Vektoren, die die Base-Editoren in das Gehirn transportieren, eine stabile Einführung von Interruptions in Hirnregionen wie Cortex und Striatum verzeichnet. Wichtig ist, dass diese Intervention nicht nur die Expansion hemmte, sondern teilweise auch zu einer Kontraktion der CAG-Repeats führte, was aus therapeutischer Sicht besonders vielversprechend ist. Sicherheitsaspekte spielen bei der Genom-Editierung eine erhebliche Rolle.
Die Analyse der Off-Target-Effekte, also unerwünschter Änderungen an anderen genomischen Stellen, zeigt, dass die Mehrheit der Baseneditionen entweder in nichtkodierenden Regionen oder als synonyme Einfügungen auftreten, die nur geringfügige oder keine Auswirkungen auf die Funktion von Proteinen haben. Dennoch bleiben potenzielle Risiken, insbesondere durch seltene nicht-synonyme Mutationen in essentiellen Genen. Daher wird intensiv an Verfeinerungen der Editoren gearbeitet, um deren Spezifität weiter zu erhöhen und die Risiken zu minimieren. Die Relevanz der Base-Editing-Technologie geht über Huntington hinaus. So konnten ähnliche Konzepte auf die Friedreich-Ataxie angewandt werden, bei der eine GAA-Repeat-Expansion im FXN-Gen die Ursache ist.
Hier werden Adenin-Base-Editoren verwendet, um gezielte Veränderungen einzuführen, die auch hier somatische Instabilität und Transkriptionsrepression vermindern. Die Therapieansätze könnten sogar den Standard von Behandlungsmöglichkeiten für eine ganze Klasse von TNR-Erkrankungen grundlegend verändern. Von fundamentaler Bedeutung ist auch die Wahl des Transportvektors für die Base-Editoren. Adeno-assoziierte Viren (AAV), speziell der Serotyp 9, bieten eine etablierte und effektive Methode, um Neuronen im zentralen Nervensystem zu erreichen. Die Verabreichung in der Neonatalphase über intrazerebroventrikuläre Injectionen hat sich für eine effiziente Verteilung bewährt.
Die andauernde Expression der Editoren ermöglicht eine langsame aber stetige Bearbeitung der Repeat-Trakte, was für den therapeutischen Erfolg essenziell ist. Die Herausforderung bleibt jedoch, auch andere wichtige Gewebe, wie Herz und gliale Zellen, die bei Huntington und Friedreich-Ataxie betroffen sind, effektiv zu erreichen. Hier könnten alternative Vektoren und Verabreichungswege die Wirksamkeit weiter verbessern. Darüber hinaus ist die Erforschung der langfristigen Auswirkungen und der potenziellen Immunantwort gegen die eingesetzten Vektoren und Editoren ein wichtiger Teil der weiteren Entwicklung. Die Jahrzehnte andauernde Forschung zur molekularen Pathogenese von Huntington und ähnlichen TNR-Erkrankungen hat gezeigt, dass nicht nur die reine Länge der Wiederholungen entscheidend ist, sondern auch deren genetische Struktur und Stabilität.